Einführung
Short Tandem Repeats (STRs), die manchmal auch als Mikrosatelliten oder Simple Sequence Repeats (SSRs) bezeichnet werden, sind ziehharmonikaähnliche DNA-Abschnitte, die Kern-Wiederholungseinheiten mit einer Länge von zwei bis sieben Nukleotiden enthalten, die sich etwa ein halbes Dutzend bis mehrere Dutzend Mal tandemartig wiederholen (1). Obwohl das menschliche Genom Tausende und Abertausende von STR-Markern enthält, wurde nur ein kleiner Kernsatz von Loci für die Verwendung in forensischen DNA- und menschlichen Identitätstests ausgewählt (2). Ähnlich wie die Verwendung einer einzigen, gemeinsamen Währung im finanziellen Sinne, ermöglichen Kernloci die gemeinsame Nutzung und den Vergleich gleichwertiger genetischer Informationen. Kommerzielle Kits sind jetzt erhältlich, um DNA-Profile zu erstellen, die diese Core STR-Loci enthalten (Tabelle 1). Millionen von STR-Profilen werden jedes Jahr weltweit von staatlichen, universitären und privaten Labors erstellt, die verschiedene Formen von menschlichen Identitätstests durchführen, einschließlich DNA-Datenbanken, forensischer Fallarbeit, Identifizierung von vermissten Personen/Massenkatastrophenopfern oder Abstammungstests.
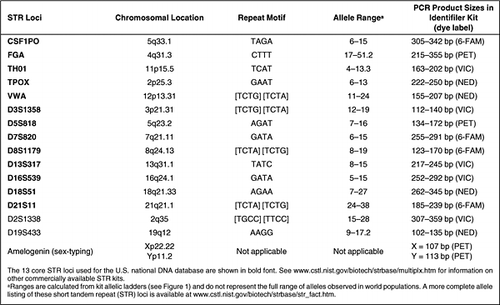
Bei der STR-Typisierung wird die PCR verwendet, um Informationen aus kleinen Mengen von verfügbarem biologischem Material zu gewinnen. Die relativ kurzen PCR-Produktgrößen von ca. 100-500 bp, die bei STR-Tests erzeugt werden, sind im Allgemeinen mit degradierter DNA kompatibel, die aufgrund von Umwelteinflüssen auf dem an einem Tatort gefundenen biologischen Beweismaterial vorhanden sein kann. Die PCR-Amplifikation mehrerer STR-Loci gleichzeitig, oder Multiplexing, ist mit verschiedenfarbigen Fluoreszenzfarbstoffen und unterschiedlich großen PCR-Produkten möglich. Die Verwendung mehrerer Loci ermöglicht eine hohe Unterscheidungskraft in einem einzigen Test, ohne viel DNA zu verbrauchen (z. B. 1 ng oder weniger an Ausgangsmaterial). Es ist erwähnenswert, dass diese Core STR-Loci zwischen Genen liegen, in denen ein hohes Maß an Variabilität toleriert wird und somit nicht direkt für körperliche Merkmale wie Haar- oder Augenfarbe oder genetische Krankheiten verantwortlich sind.
Kommerzielle Kits
Kommerziell erhältliche Kits, die vorgemischte Primer und eine Standard-Mastermischung mit Polymerase, Enzympuffern und dNTPs enthalten, vereinfachen die Erstellung von STR-Profilen und liefern Ergebnisse auf einem einheitlichen Satz von Core STR-Loci, um den nationalen und internationalen Austausch von kriminellen DNA-Profilen zu ermöglichen. Kommerzielle Kits werden in den meisten forensischen Laboratorien gegenüber In-House-Assays bevorzugt, obwohl die Kits teurer sind, da diese Kits dazu beitragen, die Verfahren zu vereinfachen und zu standardisieren und dem vielbeschäftigten Endanwender die Last der Qualitätskontrolle der PCR-Komponenten abnehmen. Darüber hinaus liefern STR-Kits Allelleitern, die häufige STR-Allele enthalten, die zuvor für die Anzahl der Wiederholungseinheiten mittels DNA-Sequenzierung charakterisiert wurden. Diese Allelleitern werden zur Kalibrierung der PCR-Produktgrößen auf die Anzahl der STR-Wiederholungen für Genotypisierungszwecke verwendet. Abbildung 1 zeigt die Allelleiter für das weit verbreitete AmpFlSTR Identifiler Kit (Applied Biosystems, Foster City, CA, USA) (3), die 205 Allele über 16 ko-amplifizierte Loci – 15 STRs und einen Amelogenin-Geschlechtstypisierungstest – enthält.
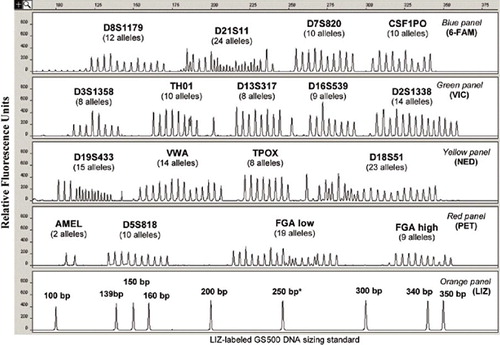
Die Genotypbestimmung in anschließend bearbeiteten Proben wird durch den Vergleich der Allelgröße (relativ zu einem internen Größenstandard) mit einer kommerziell erhältlichen STR-Kit-Allelleiter mit kalibrierter Wiederholungszahl durchgeführt, die nach demselben internen Größenstandard dimensioniert ist. Beachten Sie, dass der 250-bp-Peak im GS500-Größenstandard aufgrund von anomaler Migration typischerweise nicht verwendet wird. Nachdruck mit Genehmigung von Referenz 1, Abbildung 5.6.
Der komplette Prozess für die STR-Typisierung umfasst die Probenentnahme, die DNA-Extraktion, die DNA-Quantifizierung, die PCR-Amplifikation mehrerer STR-Loci, die STR-Allelseparierung und Größenbestimmung, die STR-Typisierung und Profilinterpretation sowie einen Bericht über die statistische Signifikanz einer Übereinstimmung (falls beobachtet). In vielen Fällen, wie z.B. bei Beweisen für sexuelle Übergriffe, können DNA-Mischungen aus einer Kombination von Körperflüssigkeiten des Opfers und des Täters resultieren und ein komplexes und schwierig zu interpretierendes Ergebnis erzeugen.
DNA-Trennungen
Nach der PCR-Amplifikation wird die Gesamtlänge des STR-Amplikons gemessen, um die Anzahl der Wiederholungen in jedem Allel im DNA-Profil zu bestimmen. Diese Längenmessung erfolgt über eine größenbasierte Trennung mittels Gel- oder Kapillarelektrophorese (CE). Jedes STR-Amplikon wurde während der PCR fluoreszenzmarkiert, da entweder der vorwärts- oder der rückwärtsgerichtete lokusspezifische Primer einen Fluoreszenzfarbstoff enthält. So kann durch Aufzeichnung der Farbstofffarbe und der Migrationszeit jedes DNA-Fragments relativ zu einem internen Größenstandard die Größe für jedes STR-Allel nach dessen Trennung von anderen STR-Allelen bestimmt werden. Zu den häufig verwendeten Geräten für die Trennung und Größenbestimmung von STR-Allelen gehören die Genanalysegeräte ABI PRISM 310 und ABI PRISM 3100 (Applied Biosystems) (4).
Es gibt eine Reihe von biologischen und instrumentellen Artefakten, die oft aussortiert werden müssen, um ein vollständiges und genaues STR-Profil zu erstellen (5; siehe auch Referenz 1, Kapitel 6 und 15). Zu den biologischen Artefakten gehören Stotterprodukte, aufgespaltene Peaks durch unvollständige Adenylierung, triallelische Muster und variante Allele, die Mutationen in den Repeat- oder flankierenden Regionen enthalten, die dazu führen, dass ein Allel außerhalb der Ladder liegt. Instrumentelle Artefakte entstehen durch Spannungsspitzen, Farbstoffflecken und Durchscheinen zwischen den Farbstoffen.
Während CE-Geräte mit Mehrfarben-Fluoreszenzdetektion, wie z. B. das ABI PRISM 3100 Genanalysegerät, derzeit das Feld dominieren, wird an der Entwicklung von Mikrochip-CE-Plattformen gearbeitet (6), um hochauflösende DNA-Trennungen mit einer möglichen Integration der PCR-Amplifikation und CE-Trennungen durchzuführen (7). Darüber hinaus wurden Massenspektrometrie (MS) mit Matrix-unterstützter Laser-Desorption/Ionisation (MALDI) und Elektrospray-Ionisation (ESI) für die STR-Typisierung ohne Allelleitern eingesetzt (8,9).
miniSTRs: Wiedergewinnung von Informationen aus degradierter DNA
DNA-Moleküle, die Wasser und/oder Hitze ausgesetzt sind, beginnen mit der Zeit, in kleinere Stücke zu zerfallen. Eine Reihe von Studien hat gezeigt, was theoretisch vorhergesagt wurde – dass DNA-Typen aus degradierten DNA-Proben effektiver wiederhergestellt werden können, wenn die PCR-Produkte kleiner sind (10-12). Indem die PCR-Primer näher an die STR-Repeat-Region gebracht werden, können die Produktgrößen reduziert werden, während die gleiche Information erhalten bleibt (11). Die Nützlichkeit von miniSTR-Assays wurde in Intra- und Interlaborstudien mit degradierten Knochenproben und gealterten Blut- und Speichelflecken bestätigt (12). Die Erfolgsraten bei der Gewinnung von Informationen aus beeinträchtigten DNA-Proben verbessern sich mit miniSTR-Systemen im Vergleich zu herkömmlichen STR-Kits. Eine Zeitleiste zur Entwicklung von miniSTRs finden Sie unter www.cstl.nist.gov/biotech/strbase/miniSTR/timeline.htm.
Auf der Grundlage ihrer 5-Farbstoff-Chemie und Mobilitätsmodifikator-Technologie hat Applied Biosystems ein miniSTR-Kit entwickelt, das in der Lage ist, acht zentrale STR-Loci und Amelogenin mit reduzierten PCR-Produktgrößen im Vergleich zu aktuellen kommerziellen Kits zu amplifizieren. Dieser AmpFlSTR MiniFiler-Kit, der eine verbesserte PCR-Mastermischung enthält, sollte die Wiederherstellung von Ergebnissen aus degradierten DNA-Proben erleichtern. Es ist jedoch wichtig zu bedenken, dass aufgrund der Verwendung anderer PCR-Primer im miniSTR-Kit im Vergleich zu früheren STR-Kits nicht übereinstimmende Ergebnisse aufgrund von Primer-Bindungsstellen-Mutationen auftreten können, die einen Allel-Dropout verursachen.
Konkordanztest
Einige STR-Allele enthalten Sequenzvariationen in den flankierenden Regionen. Wenn eine Nukleotidveränderung (oder eine Insertion oder Deletion) in einer PCR-Primer-Bindungsstelle in einem bestimmten Allel auftritt, dann ist es möglich, dass die mutierte Sequenz mit dem Primer-Annealing inkompatibel ist und das variante Allel nicht amplifiziert (1). Dieser Allel-Ausfall wird manchmal als Null-Allel bezeichnet. Eine Probe, die in Wirklichkeit heterozygot ist, kann als scheinbar homozygot erscheinen, wenn die PCR-Primer nicht annealing sind und das Allel, das die Punktmutation enthält, nicht amplifizieren (Abbildung 2). In einigen Fällen kann das Null-Allel wiederhergestellt werden, indem die Annealing-Temperatur gesenkt wird, was eine weniger strenge Bindung zwischen dem Primer und der DNA-Vorlage ermöglicht. Eine Konkordanzstudie, bei der ein Satz der gleichen DNA-Proben mit nicht überlappenden PCR-Primern getestet wird, ermöglicht die Detektion von Null-Allelen. Eine Untersuchung von MiniFiler versus Identifiler fand nur 27 Diskrepanzen bei über 10.000 verglichenen Genotypen (13). Eine Zusammenfassung der in Konkordanzstudien beobachteten diskordanten Ergebnisse ist unter www.cstl.nist.gov/biotech/strbase/NullAlleles.htm verfügbar.
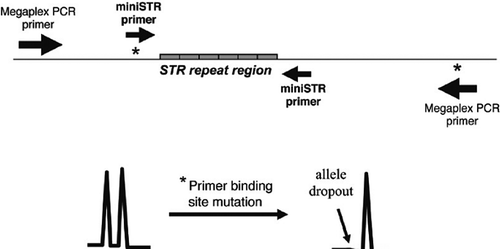
Im Allgemeinen sind die flankierenden Regionen um STR-Repeats zwischen verschiedenen Allelen konsistent, was eine robuste Amplifikation des STR-Locus ermöglicht. Die Sternchen stehen für potentielle Primer-Bindungsstellen-Mutationen, die zu Allel-Dropout und Diskordanz zwischen den getesteten Primer-Sets führen können (unteres Panel).
Solange die gleichen PCR-Primer verwendet werden, können identische STR-Typisierungsergebnisse auf der gleichen DNA-Probe erwartet werden. Werden jedoch Primerpositionen verändert, so besteht die Möglichkeit, dass Allel-Dropouts oder Größenverschiebungen auftreten. In einigen Fällen können zusätzliche Primer, die zum mutierten Allel passen – oft als degenerierte Primer bezeichnet – in die Primermischung des Kits aufgenommen werden, um eine Wiederherstellung der PCR-Amplifikation zu ermöglichen, wenn das variante Allel eine Fehlpaarung in der Primerbindungsstelle enthält (14).
Perspektiven für die Zukunft
Wie bereits vor fast 7 Jahren von der Arbeitsgruppe für Forschung und Entwicklung der National Commission on the Future of DNA Evidence (15) festgestellt wurde, werden STRs aufgrund ihrer weit verbreiteten Verwendung in nationalen DNA-Datenbanken (2,16,17) wahrscheinlich in absehbarer Zukunft die Marker der Wahl sein. Wir werden wahrscheinlich evolutionäre Fortschritte in der Typisierungstechnologie, schnellere Amplifikationsmethoden, zusätzliche STR-Kits, die neue Loci enthalten, und Computerprogramme sehen, die eine schnelle Auswertung von STR-Typisierungsdaten ermöglichen. Durch weitere Fortschritte können STR-Typisierungstechnologien miniaturisiert und mit anderen Teilen des Prozesses, wie z. B. der DNA-Extraktion und -Amplifikation, integriert werden. Durch die Erstellung von STR-Profilen mit einem gemeinsamen Satz von genetischen Markern werden heute effektive DNA-Datenbanken aufgebaut und zahlreiche forensische Fälle gelöst. Diese aktuellen Kern-Loci haben eine wichtige Rolle bei der Identitätsprüfung von Menschen gespielt und werden dies auch weiterhin tun.
Danksagungen
Der Autor, John M. Butler, wird vom National Institute of Justice durch das Interagency Agreement 2003-IJ-R-029 mit dem National Institute of Standards and Technology (NIST) Office of Law Enforcement Standards finanziert. Die Ansichten in diesem Dokument sind die des Autors und repräsentieren nicht notwendigerweise die offizielle Position oder Politik des U.S. Department of Justice. Bestimmte kommerzielle Ausrüstungen, Instrumente und Materialien sind gekennzeichnet, um experimentelle Verfahren so vollständig wie möglich zu beschreiben. In keinem Fall bedeutet eine solche Kennzeichnung eine Empfehlung oder Befürwortung durch das NIST, noch bedeutet sie, dass eines der gekennzeichneten Materialien, Instrumente oder Geräte notwendigerweise das beste für den jeweiligen Zweck ist. Kommentare zu diesem Manuskript von Peter Vallone (NIST Human Identity Project Team) wurden sehr geschätzt.
Competing Interests Statement
Der Autor erklärt keine konkurrierenden Interessen.
- 1. Butler, J.M. 2005. Forensic DNA Typing: Biology, Technology, and Genetics of STR Markers, 2nd ed. Elsevier Academic Press, New York.Google Scholar
- 2. Butler, J.M. 2006. Genetics and genomics of core STR loci used in human identity testing. J. Forensic Sci. 51:253-265.Crossref, Medline, CAS, Google Scholar
- 3. Collins, P.J., L.K. Hennessy, C.S. Leibelt, R.K. Roby, D.J. Reeder, and P.A. Foxall. 2004. Developmental validation of a singletube amplification of the 13 CODIS STR loci, D2S1338, D19S433, and amelogenin: the AmpFlSTR Identifiler PCR Amplification Kit. J. Forensic Sci. 49:1265-1277.Crossref, Medline, CAS, Google Scholar
- 4. Butler, J.M., E. Buel, F. Crivellente, and B.R. McCord. 2004. Forensic DNA typing by capillary electrophoresis: using the ABI Prism 310 and 3100 Genetic Analyzers for STR analysis. Electrophoresis 25:1397-1412.Crossref, Medline, CAS, Google Scholar
- 5. Scientific Working Group on DNA Analysis Methods (SWGDAM). 2000. Short tandem repeat (STR) interpretation guidelines. Forensic Sci. Comm. 2(3): online unter www.fbi.gov/hq/lab/fsc/backissu/july2000/strig.htm.Google Scholar
- 6. Yeung, S.H., S.A. Greenspoon, A. McGuckian, C.A. Crouse, C.A. Emrich, J. Ban, and R.A. Mathies. 2006. Schnelle und durchsatzstarke forensische Short-Tandem-Repeat-Typisierung unter Verwendung eines mikrofabrizierten Kapillar-Array-Elektrophorese-Mikrogeräts mit 96 Spuren. J. Forensic Sci. 51:740-747.Crossref, Medline, CAS, Google Scholar
- 7. Liu, P., T.S. Seo, N. Beyor, K.J. Shin, J.R. Scherer, and R.A. Mathies. 2007. Integrated portable polymerase chain reaction-capillary electrophoresis microsystem for rapid forensic short tandem repeat typing. Anal. Chem. 79:1881-1889.Crossref, Medline, CAS, Google Scholar
- 8. Butler, J.M., J. Li, T.A. Shaler, J.A. Monforte, and C.H. Becker. 1998. Reliable genotyping of short tandem repeat loci without an allelic ladder using time-of-flight mass spectrometry. Int. J. Legal Med. 112:45-49.Crossref, Medline, CAS, Google Scholar
- 9. Oberacher, H., F. Pitterl, G. Huber, H. Niederstatter, M. Steinlechner, and W. Parson. In press. Erhöhte forensische Effizienz von DNA-Fingerabdrücken durch gleichzeitige Auflösung von Längen- und Nukleotidvariabilität mittels Hochleistungsmassenspektrometrie. Hum. Mutat. Google Scholar
- 10. Wiegand, P. und M. Kleiber. 2001. Less is more-length reduction of STR amplicons using redesigned primers. Int. J. Legal Med. 114:285-287.Crossref, Medline, CAS, Google Scholar
- 11. Butler, J.M., Y. Shen, and B.R. McCord. 2003. Die Entwicklung von größenreduzierten STR-Amplikonen als Hilfsmittel für die Analyse von degradierter DNA. J. Forensic Sci. 48:1054-1064.Crossref, Medline, CAS, Google Scholar
- 12. Dixon, L.A., A.E. Dobbins, H.K. Pulker, J.M. Butler, P.M. Vallone, M.D. Coble, W. Parson, B. Berger, et al. 2006. Analyse von künstlich abgebauter DNA mittels STRs und SNPs – Ergebnisse einer europäischen Gemeinschaftsaktion (EDNAP). Forensic Sci. Int. 164:33-44.Crossref, Medline, CAS, Google Scholar
- 13. Hill, C.R., M.C. Kline, J.J. Mulero, R.E. Lagace, C.W. Chang, L.K. Hennessy, and J.M. Butler. 2007. Konkordanzstudie zwischen dem AmpFlSTR MiniFiler PCR-Amplifikationskit und herkömmlichen STR-Typisierungskits. J. Forensic Sci. 52:870-873.Crossref, Medline, CAS, Google Scholar
- 14. Leibelt, C., B. Budowle, P. Collins, Y. Daoudi, T. Moretti, G. Nunn, D. Reeder, and R. Roby. 2003. Identifizierung einer D8S1179 Primer-Bindungsstellen-Mutation und die Validierung eines Primers, der zur Wiederherstellung von Null-Allelen entwickelt wurde. Forensic Sci. Int. 133:220-227.Crossref, Medline, CAS, Google Scholar
- 15. National Institute of Justice (NIJ). 2000. The future of forensic DNA testing: predictions of the Research and Development Working Group of the National Commission on the Future of DNA Evidence, Washington, D.C. www.ojp.usdoj.gov/nij/pubs-sum/183697.htm.Google Scholar
- 16. Gill, P. 2002. Role of short tandem repeat DNA in forensic casework in the UK-past, present, and future perspectives. BioTechniques 32:366-372.Link, CAS, Google Scholar
- 17. Gill, P., D.J. Werrett, B. Budowle, and R. Guerrieri. 2004. An assessment of whether SNPs will replace STRs in national DNA databases-joint considerations of the DNA working group of the European Network of Forensic Science Institutes (ENFSI) and the Scientific Working Group on DNA Analysis Methods (SWGDAM). Sci. Justice 44:51-53.Crossref, Medline, Google Scholar