December 2010
Just How Fast Can Bacteria Grow? It Depends.
Proteomics Data Validate Model of Bacterial Growth
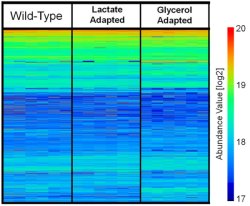
Proteomics-gegevens van PNNL stelden wetenschappers in staat om de overvloed van 900 eiwitten geïdentificeerd in wild-type E. coli in kaart te brengen in vergelijking met E. coli die was aangepast voor groei op een lactaat- of glycerolmedium. Vergroot afbeelding
Resultaten: Proteomisch onderzoek in het Pacific Northwest National Laboratory helpt bij het valideren van een bacterieel metabolisch model en geeft inzicht in de belangrijkste eiwitten en metabolische routes die essentieel zijn voor het stimuleren en ontmoedigen van bacteriële groei in een veranderende omgeving.
Waarom het belangrijk is: Bacteriën behoren tot de snelst reproducerende organismen ter wereld, met een verdubbeling elke 4 tot 20 minuten. Sommige snelgroeiende bacteriën, zoals pathogene stammen van E. coli, kunnen ons ziek maken en doden; andere bacteriën in een ondergrondse omgeving kunnen worden gebruikt om chemische verontreinigingen te immobiliseren, waardoor wordt voorkomen dat ze zich door het grondwater verplaatsen en de daarmee gepaard gaande negatieve gevolgen voor het ecosysteem. Maar of bacteriën nu schadelijk of nuttig zijn, wetenschappers moeten op betrouwbare wijze kunnen voorspellen hoe ze zich in een bepaalde omgeving zullen voortplanten en groeien.
Internationaal gerenommeerd onderzoeker Bernhard Palsson en andere onderzoekers van de Universiteit van Californië San Diego leidden een team van wetenschappers van PNNL, de Universiteit van Heidelberg en het Duitse Kankeronderzoekscentrum om te bestuderen hoe E. coli-bacteriën in een laboratoriumomgeving van de ene omgeving naar de andere veranderden en evolueerden. Het team vergeleek vervolgens proteomica, genomica en metabolische gegevens uit de studie met een computationeel model voor bacteriële groei.
Methodes: Een van de belangrijkste stukken informatie waren gegevens die werden gegenereerd met behulp van de unieke high-throughput proteomics-capaciteit van EMSL, een wetenschappelijke gebruikersfaciliteit van het Department of Energy, gevestigd in PNNL. Met behulp van high-performance vloeistofchromatografie gekoppeld aan massaspectrometrie verzamelde het team gegevens over hoe de bacteriën hun proteïneprofiel veranderden naarmate zij groeiden en zich voortplantten in verschillende mediums. De wetenschappers ontdekten dat de gegevens en het model een verrassende correlatie vertoonden (in sommige gevallen meer dan 98%), waardoor het vermogen van het model om de groei van E. coli-bacteriën onder specifieke omstandigheden te voorspellen, werd gevalideerd.
Een model maken van de groei van E. coli zal wetenschappers helpen om betrouwbare metabolische modellen te ontwikkelen voor andere, complexere organismen. Dit werk zal ook belangrijke inzichten opleveren in de beste manieren om verschillende complexe datasets te integreren om nauwkeuriger te voorspellen hoe organismen op moleculair niveau reageren op hun omgeving.
Wat is de volgende stap? De ontwikkeling van biologische modellen zal wetenschappers helpen begrijpen hoe organismen hun genetische opmaak en gen- en eiwitexpressie veranderen, zodat ingenieurs biologische organismen beter kunnen gebruiken om ons milieu en onze gezondheid te verbeteren.
Acknowledgments: Een deel van dit werk is uitgevoerd bij EMSL met gebruikmaking van capaciteiten die zijn ontwikkeld met steun van het National Institutes of Health National Center for Research Resources en het DOE Office of Biological and Environmental Research. Het werk werd ook gedeeltelijk gefinancierd door een Fulbright-beurs en subsidies van de National Science Foundation en de National Institutes of Health. Het PNNL-team bestond uit Kim Hixson, Ashoka Polpitiya, Josh Adkins, Sam Purvine, Daniel Lopez-Ferrer, en Dick Smith.