Inleiding
Korte tandemherhalingen (STR’s), die soms microsatellieten of simple sequence repeats (SSR’s) worden genoemd, zijn op een accordeon lijkende DNA-reeksen met kernrepeenheden van twee tot zeven nucleotiden lang die in tandem ongeveer een half dozijn tot enkele tientallen keren worden herhaald (1). Hoewel het menselijk genoom duizenden en duizenden STR-merkers bevat, is slechts een kleine kern van loci geselecteerd voor gebruik bij forensisch DNA-onderzoek en het testen van de menselijke identiteit (2). Net zoals het gebruik van één gemeenschappelijke munt in financiële zin, maken kernloci het mogelijk gelijkwaardige genetische informatie te delen en te vergelijken. Er zijn nu commerciële kits beschikbaar voor het genereren van DNA-profielen die deze core STR-loci bevatten (tabel 1). Elk jaar worden wereldwijd miljoenen STR-profielen gegenereerd door overheids-, universiteits- en particuliere laboratoria die verschillende vormen van menselijke identiteitstests uitvoeren, waaronder DNA-databanken, forensische zaken, identificatie van vermiste personen/massarampen of afstammingsonderzoek.
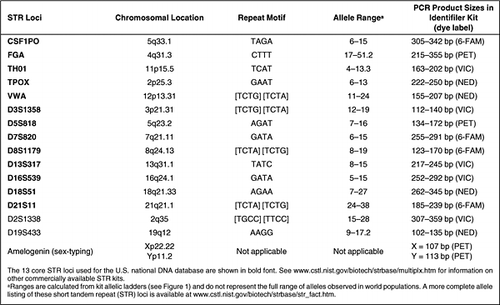
Bij STR-typering wordt PCR gebruikt om informatie te winnen uit kleine hoeveelheden beschikbaar biologisch materiaal. De relatief korte PCR-productgroottes van ongeveer 100-500 bp die met STR-testen worden gegenereerd, zijn over het algemeen compatibel met aangetast DNA dat aanwezig kan zijn als gevolg van omgevingsinvloeden op het biologisch bewijsmateriaal dat op een plaats delict is aangetroffen. PCR-amplificatie van meerdere STR-loci tegelijk, of multiplexing, is mogelijk met verschillend gekleurde fluorescerende kleurstoffen en PCR-producten van verschillende grootte. Het gebruik van meerdere loci maakt een hoog onderscheidingsvermogen in één test mogelijk zonder dat veel DNA wordt verbruikt (bv. 1 ng of minder uitgangsmateriaal). Het is vermeldenswaard dat deze STR-kernloci voorkomen tussen genen waarin een hoge mate van variabiliteit wordt getolereerd en dus niet direct verantwoordelijk zijn voor fysieke kenmerken zoals haarkleur of oogkleur of genetische ziekten.
Commerciële kits
Commercieel verkrijgbare kits met voorgemengde primers en een standaard mastermengsel met polymerase, enzymbuffers en dNTP’s vereenvoudigen het genereren van STR-profielen en leveren resultaten voor een uniforme set STR-kernloci, zodat DNA-profielen van misdadigers nationaal en internationaal kunnen worden uitgewisseld. In de meeste forensische laboratoria wordt de voorkeur gegeven aan in de handel verkrijgbare kits boven eigen tests, ook al zijn deze duurder, omdat deze kits de procedures helpen vereenvoudigen en standaardiseren en de druk bezette eindgebruiker niet langer belasten met de kwaliteitscontrole van de PCR-componenten. Bovendien leveren STR-kits allel-ladders met veel voorkomende STR-allelen waarvan het aantal herhalingseenheden vooraf via DNA-sequencing is gekarakteriseerd. Deze allel-ladders worden gebruikt om de PCR-productgrootte te kalibreren aan het aantal STR-repeereenheden voor genotyperingsdoeleinden. Figuur 1 toont de allel-ladder voor de veelgebruikte AmpFlSTR Identifiler-kit (Applied Biosystems, Foster City, CA, USA) (3) met 205 allelen over 16 gecoamplificeerde loci-15 STR’s plus een amelogenine geslachtstypebepaling.
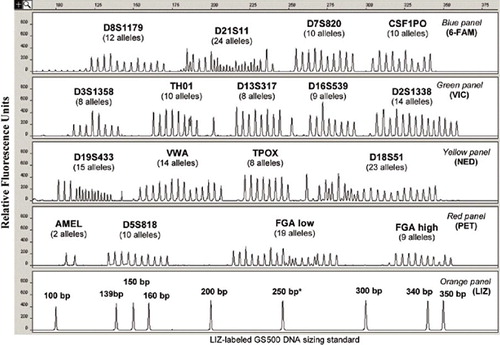
Genotypebepaling in vervolgens verwerkte monsters wordt uitgevoerd door vergelijking van de allelgrootte (ten opzichte van een interne standaard voor de grootte) met een in de handel verkrijgbare STR-kit allelische ladder met gekalibreerde herhalingsnummers, die een grootte heeft volgens dezelfde interne standaard voor de grootte. Merk op dat de piek van 250 bp in de GS500-groottestandaard gewoonlijk niet wordt gebruikt vanwege afwijkende migratie. Met toestemming overgenomen uit referentie 1, figuur 5.6.
Het volledige proces voor STR-typering omvat monstername, DNA-extractie, DNA-kwantificering, PCR-amplificatie van meerdere STR-loci, STR-allelscheiding en -dimensionering, STR-typering en profielinterpretatie, en een rapport over de statistische significantie van een overeenkomst (indien waargenomen). In veel gevallen, zoals bij aanranding, kunnen DNA-mengsels het resultaat zijn van een combinatie van lichaamsvloeistoffen van het slachtoffer en de dader.
DNA-scheidingen
Na de PCR-amplificatie wordt de totale lengte van het STR-amplicon gemeten om het aantal herhalingen te bepalen in elk allel dat in het DNA-profiel wordt aangetroffen. Deze lengtemeting wordt verricht via een scheiding op basis van grootte met behulp van gel of capillaire elektroforese (CE). Elk STR-amplicon is tijdens de PCR fluorescent gelabeld, aangezien de voorwaartse of achterwaartse locus-specifieke primer een fluorescerende kleurstof bevat. Door de kleur van de kleurstof en de migratieduur van elk DNA-fragment ten opzichte van een interne standaard te registreren, kan dus de grootte van elk STR-allel worden bepaald nadat het van andere STR-allelen is gescheiden. Veelgebruikte instrumenten voor het scheiden van STR-allelen en het bepalen van de grootte zijn de ABI PRISM 310 en ABI PRISM 3100 genetische analyzers (Applied Biosystems) (4).
Er zijn een aantal zowel biologische als instrumentele artefacten die vaak moeten worden uitgezocht om een volledig en nauwkeurig STR-profiel te genereren (5; zie ook referentie 1, hoofdstukken 6 en 15). Biologische artefacten zijn onder meer stotterproducten, gesplitste pieken door onvolledige adenylering, triallelpatronen en variante-allelen met mutaties in de repeat of flankerende regio’s waardoor een allel off-ladder is. Instrumentele artefacten ontstaan door spanningspieken, kleurstofblobs en doorschijnen tussen kleurstofkleuren.
Terwijl multikleuren fluorescentiedetectie CE-instrumenten, zoals de ABI PRISM 3100 genetic analyzer, momenteel het veld domineren, wordt gewerkt aan de ontwikkeling van microchip CE-platforms (6) voor het uitvoeren van DNA-scheidingen met hoge resolutie met uiteindelijke integratie van de PCR-amplificatie en CE-scheidingen (7). Bovendien zijn massaspectrometrie- (MS) met MALDI- (matrix-assisted laser desorption/ionization) en ESI- (electrospray ionization) technieken gebruikt voor STR-typering zonder allelische ladders (8,9).
miniSTRs: Recovery of Information From Degraded DNA
DNA-moleculen die aan water en/of warmte worden blootgesteld, zullen na verloop van tijd in kleinere stukjes uiteenvallen. Uit een aantal studies is gebleken wat theoretisch voorspeld wordt, namelijk dat DNA-types doeltreffender uit afgebroken DNA-monsters kunnen worden teruggewonnen wanneer de PCR-producten kleiner zijn (10-12). Door de PCR-primers dichter bij het STR-repeatgebied te plaatsen, kan de productgrootte worden verkleind met behoud van dezelfde informatie (11). De bruikbaarheid van miniSTR-tests is bevestigd in intra- en interlaboratoriumstudies met aangetaste botmonsters en verouderde bloed- en speekselvlekken (12). De succespercentages bij het terugwinnen van informatie uit aangetaste DNA-monsters verbeteren met miniSTR-systemen in vergelijking met conventionele STR-kits. Een tijdlijn voor de ontwikkeling van miniSTRs is te vinden op www.cstl.nist.gov/biotech/strbase/miniSTR/timeline.htm.
Met gebruikmaking van hun 5-dye chemie en mobiliteitsmodificatietechnologie heeft Applied Biosystems een miniSTR-kit ontwikkeld waarmee acht core STR-loci en amelogenine kunnen worden geamplificeerd met een kleinere PCR-productgrootte dan met de huidige commerciële kits. Deze AmpFlSTR MiniFiler-kit, die een verbeterd PCR-mastermengsel bevat, moet helpen bij de inspanningen om resultaten van gedegradeerde DNA-monsters terug te krijgen. Het is echter belangrijk in gedachten te houden dat, omdat bij de miniSTR-kit andere PCR-primers worden gebruikt dan bij eerdere STR-kits, er discordante resultaten kunnen optreden als gevolg van primerbindingsplaatsmutaties die alleluitval veroorzaken.
Concordantietests
Sommige STR-allelen bevatten sequentievariatie in de flankerende regio’s. Als een nucleotideverandering (of insertie of deletie) optreedt in een PCR-primerbindingsplaats in een bepaald allel, is het mogelijk dat de gemuteerde sequentie incompatibel is met de annealing van de primer en dat het allelvariant niet amplificeert (1). Deze uitval van het allel wordt ook wel een null-allel genoemd. Een monster dat eigenlijk een heterozygoot is, kan als een schijnbare homozygoot tevoorschijn komen als de PCR-primers er niet in slagen het allel dat de puntmutatie bevat te annealiseren en te amplificeren (figuur 2). In sommige gevallen kan het nulde allel worden teruggevonden door de annealingtemperatuur te verlagen, waardoor een minder stringente binding tussen de primer en het DNA-sjabloon mogelijk wordt. Een concordantiestudie, waarbij een reeks van dezelfde DNA-monsters met niet-overlappende PCR-primers wordt getest, maakt detectie van nulde allelen mogelijk. Bij een onderzoek van MiniFiler versus Identifiler werden slechts 27 discrepanties gevonden in meer dan 10.000 vergeleken genotypes (13). Een samenvatting van discordante resultaten die in concordantiestudies zijn waargenomen, is beschikbaar op www.cstl.nist.gov/biotech/strbase/NullAlleles.htm.
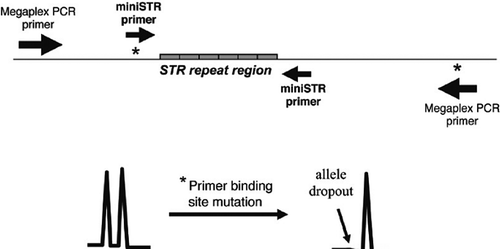
De flankerende regio’s rond STR-repeats zijn over het algemeen consistent tussen verschillende allelen, waardoor robuuste amplificatie van de STR-locus mogelijk is. De sterretjes staan voor potentiële primerbindingsplaatsmutaties die kunnen leiden tot alleluitval en discordantie tussen de geteste primersets (onderste paneel).
Zolang dezelfde PCR-primers worden gebruikt, kunnen identieke STR-typeringsresultaten worden verwacht op hetzelfde DNA-monster. Als de primerposities echter worden gewijzigd, bestaat de mogelijkheid van allel drop-out of grootteverschuiving. In sommige gevallen kunnen extra primers die overeenkomen met het gemuteerde allel – vaak degenerate primers genoemd – in het primermengsel van de kit worden opgenomen om herstel van PCR-amplificatie mogelijk te maken wanneer het variant-allel met een mismatch in de primerbindingsplaats aanwezig is (14).
Perspectieven voor de toekomst
Zoals bijna 7 jaar geleden werd opgemerkt door de Werkgroep Onderzoek en Ontwikkeling van de Nationale Commissie voor de toekomst van DNA-bewijs (15), zullen STR’s in de nabije toekomst waarschijnlijk de favoriete merkers blijven, omdat zij op grote schaal worden gebruikt in nationale DNA-databanken (2,16,17). We zullen waarschijnlijk te maken krijgen met evolutionaire vooruitgang in de typeringstechnologie, snellere amplificatiemethoden, extra STR-kits die nieuwe loci bevatten, en computerprogramma’s die een snelle evaluatie van STR-typeringsgegevens mogelijk maken. Door voortdurende vooruitgang kunnen STR-typeringstechnologieën worden geminiaturiseerd en worden geïntegreerd met andere onderdelen van het proces, zoals DNA-extractie en amplificatie. Er worden doeltreffende DNA-databanken aangelegd en talrijke forensische zaken worden vandaag opgelost door het genereren van STR-profielen met een gemeenschappelijke reeks genetische markers. Deze huidige core loci hebben een vitale rol gespeeld bij het testen van de identiteit van mensen en zullen dat ook in de toekomst blijven doen.
Acknowledgments
De auteur, John M. Butler, wordt gefinancierd door het National Institute of Justice via interagency agreement 2003-IJ-R-029 met het National Institute of Standards and Technology (NIST) Office of Law Enforcement Standards. De standpunten in dit document zijn die van de auteur en geven niet noodzakelijkerwijs het officiële standpunt of beleid van het Amerikaanse ministerie van Justitie weer. Bepaalde commerciële apparatuur, instrumenten en materialen worden genoemd om de experimentele procedures zo volledig mogelijk te beschrijven. In geen geval impliceert een dergelijke identificatie een aanbeveling of goedkeuring door het NIST, noch impliceert dit dat de geïdentificeerde materialen, instrumenten of apparatuur noodzakelijkerwijs de beste zijn die voor het doel beschikbaar zijn. Commentaar op dit manuscript van Peter Vallone (NIST Human Identity Project Team) werd zeer op prijs gesteld.
Competing Interests Statement
De auteur verklaart geen concurrerende belangen te hebben.
- 1. Butler, J.M. 2005. Forensische DNA-typering: Biology, Technology, and Genetics of STR Markers, 2nd ed. Elsevier Academic Press, New York.Google Scholar
- 2. Butler, J.M. 2006. Genetics and genomics of core STR loci used in human identity testing. J. Forensic Sci. 51:253-265.Crossref, Medline, CAS, Google Scholar
- 3. Collins, P.J., L.K. Hennessy, C.S. Leibelt, R.K. Roby, D.J. Reeder, and P.A. Foxall. 2004. Developmental validation of a singletube amplification of the 13 CODIS STR loci, D2S1338, D19S433, and amelogenin: the AmpFlSTR Identifiler PCR Amplification Kit. J. Forensic Sci. 49:1265-1277.Crossref, Medline, CAS, Google Scholar
- 4. Butler, J.M., E. Buel, F. Crivellente, and B.R. McCord. 2004. Forensic DNA typing by capillary electrophoresis: using the ABI Prism 310 and 3100 Genetic Analyzers for STR analysis. Electrophoresis 25:1397-1412.Crossref, Medline, CAS, Google Scholar
- 5. Wetenschappelijke Werkgroep DNA-analysemethoden (SWGDAM). 2000. Richtlijnen voor de interpretatie van short tandem repeat (STR). Forensic Sci. Comm. 2(3): online op www.fbi.gov/hq/lab/fsc/backissu/july2000/strig.htm.Google Scholar
- 6. Yeung, S.H., S.A. Greenspoon, A. McGuckian, C.A. Crouse, C.A. Emrich, J. Ban, and R.A. Mathies. 2006. Rapid and high-throughput forensic short tandem repeat typing using a 96-lane microfabricated capillary array electrophoresis microdevice. J. Forensic Sci. 51:740-747.Crossref, Medline, CAS, Google Scholar
- 7. Liu, P., T.S. Seo, N. Beyor, K.J. Shin, J.R. Scherer, and R.A. Mathies. 2007. Integrated portable polymerase chain reaction-capillary electrophoresis microsystem for rapid forensic short tandem repeat typing. Anal. Chem. 79:1881-1889.Crossref, Medline, CAS, Google Scholar
- 8. Butler, J.M., J. Li, T.A. Shaler, J.A. Monforte, and C.H. Becker. 1998. Reliable genotyping of short tandem repeat loci without an allelic ladder using time-of-flight mass spectrometry. Int. J. Legal Med. 112:45-49.Crossref, Medline, CAS, Google Scholar
- 9. Oberacher, H., F. Pitterl, G. Huber, H. Niederstatter, M. Steinlechner, and W. Parson. In press. Increased forensic efficiency of DNA fingerprints through simultaneous resolution of length and nucleotide variability by high-performance mass spectrometry. Hum. Mutat. Google Scholar
- 10. Wiegand, P. en M. Kleiber. 2001. Less is more-length reduction of STR amplicons using redesigned primers. Int. J. Legal Med. 114:285-287.Crossref, Medline, CAS, Google Scholar
- 11. Butler, J.M., Y. Shen, and B.R. McCord. 2003. The development of reduced size STR amplicons as tools for analysis of degraded DNA. J. Forensic Sci. 48:1054-1064.Crossref, Medline, CAS, Google Scholar
- 12. Dixon, L.A., A.E. Dobbins, H.K. Pulker, J.M. Butler, P.M. Vallone, M.D. Coble, W. Parson, B. Berger, et al. 2006. Analyse van kunstmatig aangetast DNA met behulp van STR’s en SNP’s – resultaten van een gezamenlijke Europese oefening (EDNAP). Forensic Sci. Int. 164:33-44.Crossref, Medline, CAS, Google Scholar
- 13. Hill, C.R., M.C. Kline, J.J. Mulero, R.E. Lagace, C.W. Chang, L.K. Hennessy, and J.M. Butler. 2007. Concordantiestudie tussen de AmpFlSTR MiniFiler PCR amplificatiekit en conventionele STR-typeringskits. J. Forensic Sci. 52:870-873.Crossref, Medline, CAS, Google Scholar
- 14. Leibelt, C., B. Budowle, P. Collins, Y. Daoudi, T. Moretti, G. Nunn, D. Reeder, and R. Roby. 2003. Identification of a D8S1179 primer binding site mutation and the validation of a primer designed to recover null alleles. Forensic Sci. Int. 133:220-227.Crossref, Medline, CAS, Google Scholar
- 15. National Institute of Justice (NIJ). 2000. The future of forensic DNA testing: predictions of the Research and Development Working Group of the National Commission on the Future of DNA Evidence, Washington, D.C. www.ojp.usdoj.gov/nij/pubs-sum/183697.htm.Google Scholar
- 16. Gill, P. 2002. Role of short tandem repeat DNA in forensic casework in the UK-past, present, and future perspectives. BioTechniques 32:366-372.Link, CAS, Google Scholar
- 17. Gill, P., D.J. Werrett, B. Budowle, and R. Guerrieri. 2004. An assessment of SNPs will replace STRs in national DNA databases-joint considerations of the DNA working group of the European Network of Forensic Science Institutes (ENFSI) and the Scientific Working Group on DNA Analysis Methods (SWGDAM). Sci. Justice 44:51-53.Crossref, Medline, Google Scholar