Introdução
Repetições tandem curtas (STRs), que são por vezes referidas como microsatélites ou repetições de sequência simples (SSRs), são trechos de ADN do tipo acordeão contendo unidades de repetição de núcleo de entre dois e sete nucleótidos de comprimento que são repetidas em tandem de aproximadamente meia dúzia a várias dúzias de vezes (1). Embora o genoma humano contenha milhares e milhares de marcadores STR, apenas um pequeno núcleo de loci foi seleccionado para utilização em testes forenses de ADN e identidade humana (2). Tal como a utilização de uma moeda única e comum no sentido financeiro, os loci centrais permitem a partilha e comparação de informação genética equivalente. Os kits comerciais estão agora disponíveis para gerar perfis de ADN contendo estes loci STR essenciais (Quadro 1). Milhões de perfis STR são gerados em todo o mundo todos os anos por laboratórios governamentais, universitários e privados que realizam várias formas de testes de identidade humana, incluindo banco de dados de ADN, trabalho de investigação forense, identificação de pessoas desaparecidas/vítimas de desastres de massa, ou testes de parentesco.
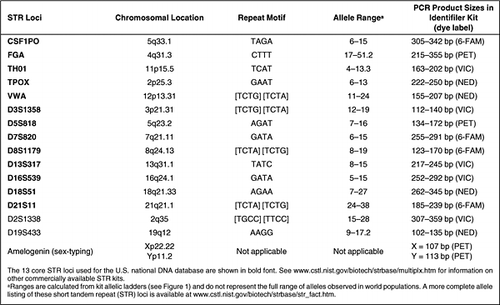
caption>Table 1. Características dos 15 STR Loci Presentes no Kit Comercialmente Disponível AmpFlSTR Identifiler
>br>
Com a tipagem STR, a PCR é utilizada para recuperar informação a partir de pequenas quantidades de material biológico disponível. Os tamanhos relativamente curtos dos produtos PCR de aproximadamente 100-500 bp gerados com testes STR são geralmente compatíveis com ADN degradado que pode estar presente devido a insultos ambientais sobre o material biológico probatório encontrado no local do crime. A amplificação por PCR de múltiplos loci STR simultaneamente, ou multiplexagem, é possível com corantes fluorescentes de diferentes cores e produtos de PCR de diferentes tamanhos. A utilização de múltiplos loci permite um elevado poder de discriminação num único teste sem consumir muito ADN (por exemplo, 1 ng ou menos de material de base). Vale a pena notar que estes loci STR centrais ocorrem entre genes em que um elevado grau de variabilidade é tolerado e não são, portanto, directamente responsáveis por traços físicos como a cor do cabelo ou a cor dos olhos ou doenças genéticas.
Kits comerciais
Kits disponíveis comercialmente, que fornecem primers pré-misturados e uma mistura principal padrão contendo a polimerase, tampões enzimáticos, e dNTPs, simplificam a geração de perfis STR e fornecem resultados sobre um conjunto uniforme de núcleos de STR loci para possibilitar a partilha nacional e internacional de perfis de DNA criminal. Os kits comerciais são preferidos na maioria dos laboratórios forenses em vez dos ensaios internos, embora os kits sejam mais caros, uma vez que estes kits ajudam a simplificar e padronizar os procedimentos e a remover o fardo do controlo de qualidade dos componentes PCR do utilizador final ocupado. Além disso, os kits STR fornecem escadas alélicas contendo alelos STR comuns que foram previamente caracterizados pelo número de unidades repetidas através de sequenciação de ADN. Estas escadas alélicas são utilizadas para calibrar os tamanhos de produtos PCR para o número de repetição STR para fins de genotipagem. A figura 1 mostra a escada alélica para o kit de identificação AmpFlSTR amplamente utilizado (Applied Biosystems, Foster City, CA, EUA) (3) contendo 205 alelos em 16 loci-15 STRs coamplificados mais um ensaio de tipagem sexual de amelogenina.
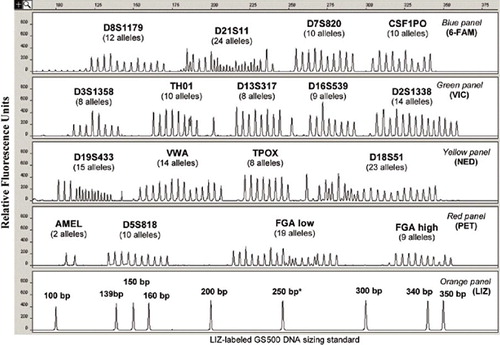
A determinação do genótipo em amostras processadas subsequentemente é realizada comparando o tamanho do alelo (em relação a um padrão de tamanho interno) com um kit de escada alélica STR fornecido comercialmente com números de repetição calibrados, que é dimensionado de acordo com o mesmo padrão de tamanho interno. Note-se que o pico de 250-bp no padrão de tamanho GS500 não é tipicamente utilizado devido a uma migração anómala. Reimpresso com permissão da Referência 1, Figura 5.6.
O processo completo de tipagem STR inclui a recolha de amostras, extracção de ADN, quantificação de ADN, amplificação PCR de múltiplos loci STR, separação e dimensionamento de alelos STR, tipagem STR e interpretação de perfis, e um relatório do significado estatístico de uma correspondência (se observado). Em muitas situações de trabalho de caso, tais como provas de agressão sexual, as misturas de ADN podem resultar de uma combinação dos fluidos corporais da vítima e do perpetrador e criar um resultado complexo e desafiante para a interpretação.
Separações de ADN
Aplicação da PCR subsequente, o comprimento total do amplicon STR é medido para determinar o número de repetições presentes em cada alelo encontrado no perfil de ADN. Esta medição do comprimento é feita através de uma separação com base no tamanho envolvendo gel ou electroforese capilar (CE). Cada amplicon STR foi rotulado fluorescentemente durante a PCR, uma vez que ou o primer específico do locus para a frente ou para trás contém um corante fluorescente. Assim, ao registar a cor do corante e o tempo de migração de cada fragmento de ADN em relação a um padrão de tamanho interno, o tamanho para cada alelo STR pode ser determinado após a sua separação de outros alelos STR. Os instrumentos comummente utilizados para a separação e dimensionamento dos alelos STR incluem os analisadores genéticos ABI PRISM 310 e ABI PRISM 3100 (Applied Biosystems) (4).
Existe um número de artefactos biológicos e instrumentais que muitas vezes têm de ser classificados a fim de gerar um perfil STR completo e preciso (5; ver também Referência 1, Capítulos 6 e 15). Os artefactos biológicos incluem produtos com gaguez, picos divididos de adenilação incompleta, padrões trialleicos, e alelos variantes contendo mutações nas regiões de repetição ou de flanco que causam um alelo fora da bexiga. Os artefactos instrumentais surgem de picos de tensão, manchas de corante, e hemorragia entre cores de corante.
Embora os instrumentos de detecção de fluorescência multicolorida CE, tais como o analisador genético ABI PRISM 3100, dominam actualmente o campo, estão em curso esforços para desenvolver plataformas de microchip CE (6) para realizar separações de ADN de alta resolução com eventual integração da amplificação PCR e separações CE (7). Além disso, foram utilizadas técnicas de espectrometria de massa (MS) com dessorção/ionização laser assistida por matriz (MALDI) e ionização por electrospray (ESI) para a tipagem STR sem escadas alélicas (8,9).
miniSTRs: Recuperação de informação de ADN degradado
moléculas de ADN que são expostas à água e/ou calor começarão, com o tempo, a decompor-se em pedaços mais pequenos. Vários estudos mostraram o que é teoricamente previsto – que os tipos de ADN podem ser recuperados mais eficazmente a partir de amostras de ADN degradado quando os produtos da PCR são mais pequenos (10-12). Ao aproximar os iniciadores de PCR da região de repetição STR, os tamanhos dos produtos podem ser reduzidos, mantendo a mesma informação (11). A utilidade dos ensaios de miniSTR foi confirmada em estudos intra e interlaboratoriais envolvendo amostras ósseas degradadas e manchas de sangue e saliva envelhecidas (12). As taxas de sucesso na recuperação de informação de amostras de ADN comprometidas melhoram com os sistemas miniSTR em comparação com os kits STR convencionais. Uma linha temporal cobrindo o desenvolvimento de miniSTR pode ser encontrada em www.cstl.nist.gov/biotech/strbase/miniSTR/timeline.htm.
Usando a sua química de 5 tinturas e tecnologia modificadora de mobilidade, a Applied Biosystems desenvolveu um kit miniSTR capaz de amplificar oito loci STR de núcleo e amelogenina com tamanhos reduzidos de produtos PCR relativamente aos actuais kits comerciais. Este kit AmpFlSTR MiniFiler, que inclui uma mistura mestre de PCR melhorada, deverá ajudar nos esforços de recuperação de resultados de amostras de ADN degradadas. No entanto, é importante ter em mente que, uma vez que estão em uso diferentes primários de PCR com o kit miniSTR relativamente a kits STR anteriores, podem ocorrer resultados discordantes devido a mutações do local de ligação dos primários que causam a queda dos alelos.
Testes de concordância
alguns alelos STR contêm variação de sequência nas regiões de flanco. Se ocorrer uma alteração do nucleótido (ou inserção ou eliminação) num local de ligação de primer PCR num determinado alelo, então é possível que a sequência mutante seja incompatível com o recozimento de primer e o alelo variante não conseguirá amplificar (1). Esta desistência alélica é por vezes referida como um alelo nulo. Uma amostra que é realmente um heterozigoto pode aparecer como um homozigoto aparente se os iniciadores da PCR não conseguirem recozir e amplificar o alelo que contém a mutação pontual (Figura 2). Em alguns casos, o alelo nulo pode ser recuperado através da redução da temperatura de recozimento, permitindo uma ligação menos rigorosa entre o primário e o modelo de ADN. Um estudo de concordância, que testa um conjunto das mesmas amostras de ADN com primers PCR não sobrepostos, permite a detecção de alelos nulos. Um exame do MiniFiler versus Identificfiler encontrou apenas 27 discrepâncias em mais de 10.000 genótipos em comparação (13). Um resumo dos resultados discordantes observados nos estudos de concordância está disponível em www.cstl.nist.gov/biotech/strbase/NullAlleles.htm.
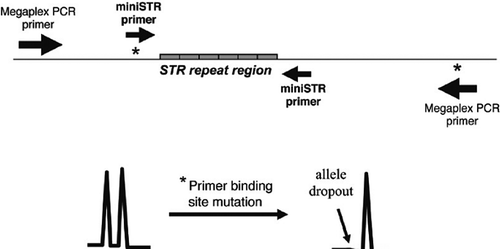
Geralmente, as regiões de flanco em torno das repetições STR são consistentes entre vários alelos, permitindo uma amplificação robusta do locus STR. Os asteriscos representam potenciais mutações do local de ligação do primário que poderiam levar à desistência de alelos e à discordância entre os conjuntos de primários testados (painel inferior).
Desde que sejam utilizados os mesmos primários de PCR, podem esperar-se resultados idênticos de tipagem de STR na mesma amostra de ADN. No entanto, se as posições dos iniciadores forem alteradas, então existe a possibilidade de ocorrer a desistência do alelo ou mudança de tamanho. Em alguns casos, os iniciadores extra correspondentes ao alelo mutante – frequentemente referidos como iniciadores degenerados – podem ser incluídos na mistura de iniciadores do kit para permitir a recuperação da amplificação da PCR quando o alelo variante contendo um desajuste no local de ligação do iniciador estiver presente (14).
Perspectivas sobre o Futuro
Como observado há quase 7 anos pelo Grupo de Trabalho de Investigação e Desenvolvimento da Comissão Nacional sobre o Futuro das Evidências de ADN (15), os STR serão provavelmente os marcadores de escolha para o futuro previsível devido à sua utilização generalizada nas bases de dados nacionais de ADN (2,16,17). Veremos provavelmente uma progressão evolutiva na tecnologia de tipagem, métodos de amplificação mais rápidos, kits STR adicionais contendo novos loci, e programas informáticos que permitirão uma avaliação rápida dos dados de tipagem STR. Através de avanços contínuos, as tecnologias de tipagem STR podem tornar-se miniaturizadas e integradas com outras partes do processo, tais como a extracção e amplificação de ADN. Bases de dados de ADN eficazes estão a ser construídas e numerosos casos forenses resolvidos actualmente através da geração de perfis STR com um conjunto comum de marcadores genéticos. Estes loci centrais actuais têm desempenhado e continuarão a desempenhar um papel vital nos testes de identidade humana.
Avalores
O autor, John M. Butler, é financiado pelo Instituto Nacional de Justiça através de um acordo inter-agências 2003-IJ-R-029 com o Instituto Nacional de Normas e Tecnologia (NIST) Office of Law Enforcement Standards. Os pontos de vista neste documento são os do autor e não representam necessariamente a posição ou políticas oficiais do Departamento de Justiça dos Estados Unidos. Certos equipamentos, instrumentos e materiais comerciais são identificados para especificar os procedimentos experimentais da forma mais completa possível. Em nenhum caso tal identificação implica uma recomendação ou endosso do NIST nem implica que qualquer dos materiais, instrumentos ou equipamentos identificados sejam necessariamente os melhores disponíveis para o efeito. Os comentários sobre este manuscrito por Peter Vallone (NIST Human Identity Project Team) foram muito apreciados.
Competing Interests Statement
O autor declara não existirem interesses concorrentes.
- 1. Butler, J.M. 2005. Dactilografia de DNA Forense: Biology, Technology, and Genetics of STR Markers, 2ª ed. Elsevier Academic Press, New York.Google Scholar
- 2. Butler, J.M. 2006. Genetics and genomics of core STR loci used in human identity testing. J. Forensic Sci. 51:253-265.Crossref, Medline, CAS, Google Scholar
- 3. Collins, P.J., L.K. Hennessy, C.S. Leibelt, R.K. Roby, D.J. Reeder, e P.A. Foxall. 2004. Validação do desenvolvimento de uma amplificação de tubo único dos 13 CODIS STR loci, D2S1338, D19S433, e amelogenina: o AmpFlSTR Identifiler PCR Amplification Kit. J. Forensic Sci. 49:1265-1277.Crossref, Medline, CAS, Google Scholar
- 4. Butler, J.M., E. Buel, F. Crivellente, e B.R. McCord. 2004. Tipagem forense de ADN por electroforese capilar: utilizando os analisadores genéticos ABI Prism 310 e 3100 para análise STR. Electroforese 25:1397-1412.Crossref, Medline, CAS, Google Scholar
- 5. Grupo de Trabalho Científico sobre Métodos de Análise de ADN (SWGDAM). 2000. Short tandem repeat (STR) interpretation guidelines. Forensic Sci. Comm. 2(3): online em www.fbi.gov/hq/lab/fsc/backissu/july2000/strig.htm.Google Scholar
- 6. Yeung, S.H., S.A. Greenspoon, A. McGuckian, C.A. Crouse, C.A. Emrich, J. Ban, e R.A. Mathies. 2006. Rápida e de alto rendimento, repetição de digitação forense em tandem de curta duração usando um microdispositivo de eletroforese de matriz capilar de 96 pistas. J. Forensic Sci. 51:740-747.Crossref, Medline, CAS, Google Scholar
- 7. Liu, P., T.S. Seo, N. Beyor, K.J. Shin, J.R. Scherer, e R.A. Mathies. 2007. Microssistema portátil integrado de electroforese de reacção em cadeia da polimerase para repetição rápida da tipagem forense em tandem curto. Anal. Química. 79:1881-1889.Crossref, Medline, CAS, Google Scholar
- 8. Butler, J.M., J. Li, T.A. Shaler, J.A. Monforte, e C.H. Becker. 1998. Genotipagem fiável de loci de repetição em tandem curto sem escada alélica usando espectrometria de massa de tempo de voo. Int. J. Med. Legal. 112:45-49.Crossref, Medline, CAS, Google Scholar
- 9. Oberacher, H., F. Pitterl, G. Huber, H. Niederstatter, M. Steinlechner, e W. Parson. No prelo. Aumento da eficiência forense das impressões digitais de ADN através da resolução simultânea da variabilidade do comprimento e dos nucleótidos por espectrometria de massa de alto desempenho. Hum. Mutat. Google Scholar
- 10. Wiegand, P. e M. Kleiber. 2001. Less is more-length reduction of STR amplicons using redesigned primers. Int. J. Med. Legal. 114:285-287.Crossref, Medline, CAS, Google Scholar
- 11. Butler, J.M., Y. Shen, e B.R. McCord. 2003. O desenvolvimento de amplicons STR de tamanho reduzido como ferramentas para análise de ADN degradado. J. Forensic Sci. 48:1054-1064.Crossref, Medline, CAS, Google Scholar
- 12. Dixon, L.A., A.E. Dobbins, H.K. Pulker, J.M. Butler, P.M. Vallone, M.D. Coble, W. Parson, B. Berger, et al. 2006. Análise de ADN artificialmente degradado usando STRs e SNPs-resultados de um exercício europeu colaborativo (EDNAP). Forensic Sci. Int. 164:33-44.Crossref, Medline, CAS, Google Scholar
- 13. Hill, C.R., M.C. Kline, J.J. Mulero, R.E. Lagace, C.W. Chang, L.K. Hennessy, e J.M. Butler. 2007. Estudo de concordância entre o kit de amplificação AmpFlSTR MiniFiler PCR e os kits de tipagem STR convencionais. J. Forensic Sci. 52:870-873.Crossref, Medline, CAS, Google Scholar
- 14. Leibelt, C., B. Budowle, P. Collins, Y. Daoudi, T. Moretti, G. Nunn, D. Reeder, e R. Roby. 2003. Identificação de uma mutação do local de ligação de um primário D8S1179 e validação de um primário concebido para recuperar alelos nulos. Forensic Sci. Int. 133:220-227.Crossref, Medline, CAS, Google Scholar
- 15. Instituto Nacional de Justiça (NIJ). 2000. The future of forensic DNA testing: predictions of the Research and Development Working Group of the National Commission on the Future of DNA Evidence, Washington, D.C. www.ojp.usdoj.gov/nij/pubs-sum/183697.htm.Google Scholar
- 16. Gill, P. 2002. Papel da repetição de ADN em tandem curto em casos forenses no Reino Unido – perspectivas passadas, presentes, e futuras. BioTechniques 32:366-372.Link, CAS, Google Scholar
- 17. Gill, P., D.J. Werrett, B. Budowle, e R. Guerrieri. 2004. Uma avaliação sobre se os SNPs irão substituir os STRs nas bases de dados nacionais de ADN – considerações conjuntas do grupo de trabalho de ADN da Rede Europeia de Institutos de Ciência Forense (ENFSI) e do Grupo de Trabalho Científico sobre Métodos de Análise de ADN (SWGDAM). Sci. Justice 44:51-53.Crossref, Medline, Google Scholar