Diciembre de 2010
¿Cuán rápido pueden crecer las bacterias? Depende.
Los datos proteómicos validan el modelo de crecimiento bacteriano
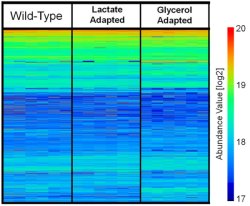
Los datos proteómicos del PNNL permitieron a los científicos trazar un mapa de la abundancia de 900 proteínas identificadas en la E. coli de tipo salvaje en comparación con la E. coli que había sido adaptada para crecer en medios con lactato o glicerol. Ampliar la imagen
Resultados: La investigación proteómica del Laboratorio Nacional del Noroeste del Pacífico está ayudando a validar un modelo metabólico bacteriano y está proporcionando conocimientos sobre las proteínas clave y las vías metabólicas esenciales para fomentar y desalentar el crecimiento bacteriano en un entorno cambiante.
Por qué es importante: Las bacterias se encuentran entre los organismos de reproducción más rápida del mundo, duplicándose cada 4 a 20 minutos. Algunas bacterias de crecimiento rápido, como las cepas patógenas de E. coli, pueden enfermar y matarnos; otras bacterias en un entorno subterráneo pueden servir para inmovilizar contaminantes químicos, impidiendo su movimiento a través de las aguas subterráneas y cualquier impacto negativo asociado en el ecosistema. Pero tanto si las bacterias son perjudiciales como si son útiles, los científicos tienen que ser capaces de predecir de forma fiable cómo se reproducirán y crecerán en un entorno concreto.
El investigador de renombre internacional Bernhard Palsson y otros investigadores de la Universidad de California en San Diego dirigieron un equipo de científicos del PNNL, la Universidad de Heidelberg y el Centro Alemán de Investigación del Cáncer para estudiar cómo las bacterias E. coli cambiaban y evolucionaban de un entorno a otro en un entorno de laboratorio. A continuación, el equipo comparó los datos proteómicos, genómicos y metabólicos del estudio con un modelo computacional de crecimiento bacteriano.
Métodos: Una de las piezas clave de información fueron los datos generados mediante la capacidad única de proteómica de alto rendimiento del EMSL, una instalación de usuarios científicos del Departamento de Energía situada en el PNNL. Utilizando la cromatografía líquida de alto rendimiento acoplada a la espectrometría de masas, el equipo recopiló datos sobre cómo las bacterias cambiaban su perfil proteico a medida que crecían y se reproducían en diferentes medios. Los científicos descubrieron que los datos y el modelo estaban correlacionados en un grado sorprendente (más del 98% en algunos casos), validando la capacidad del modelo para predecir el crecimiento de la bacteria E. coli en condiciones específicas.
Ser capaz de modelar el crecimiento de E. coli ayudará a los científicos a empezar a desarrollar modelos metabólicos fiables para otros organismos más complejos. Este trabajo también proporcionará conocimientos clave sobre las mejores formas de integrar diferentes conjuntos de datos complejos para predecir con mayor precisión cómo responden los organismos a sus entornos a nivel molecular.
Lo que sigue: El desarrollo de modelos biológicos ayudará a los científicos a comprender cómo los organismos cambian su composición genética y la expresión de genes y proteínas, de modo que los ingenieros puedan utilizar mejor los organismos biológicos para mejorar nuestro entorno y nuestra salud.
Agradecimientos: Parte de este trabajo se realizó en el EMSL utilizando capacidades desarrolladas con el apoyo del Centro Nacional de Recursos de Investigación de los Institutos Nacionales de Salud y la Oficina de Investigación Biológica y Ambiental del DOE. El trabajo también fue financiado en parte por una beca Fulbright y subvenciones de la Fundación Nacional de la Ciencia y los Institutos Nacionales de la Salud. El equipo del PNNL estaba formado por Kim Hixson, Ashoka Polpitiya, Josh Adkins, Sam Purvine, Daniel López-Ferrer y Dick Smith.