Introducción
Las repeticiones cortas en tándem (STRs), que a veces se denominan microsatélites o repeticiones de secuencia simple (SSRs), son tramos de ADN en forma de acordeón que contienen unidades de repetición del núcleo de entre dos y siete nucleótidos de longitud que se repiten en tándem desde aproximadamente media docena hasta varias docenas de veces (1). Aunque el genoma humano contiene miles y miles de marcadores de STR, sólo se ha seleccionado un pequeño conjunto de loci para su uso en pruebas forenses de ADN y de identidad humana (2). Al igual que el uso de una moneda única y común en un sentido financiero, los loci centrales permiten compartir y comparar información genética equivalente. En la actualidad existen kits comerciales para generar perfiles de ADN que contienen estos loci STR básicos (Tabla 1). Cada año se generan millones de perfiles de STR en todo el mundo por parte de laboratorios gubernamentales, universitarios y privados que realizan diversas formas de pruebas de identidad humana, como la creación de bases de datos de ADN, los casos forenses, la identificación de personas desaparecidas/víctimas de catástrofes masivas o las pruebas de filiación.
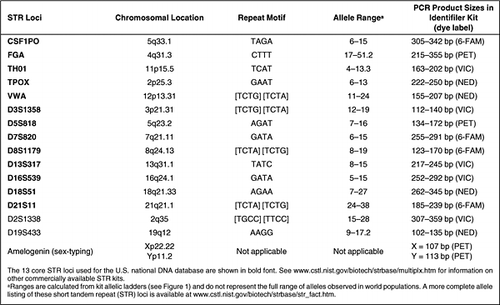
Con la tipificación de STR, la PCR se utiliza para recuperar información de pequeñas cantidades de material biológico disponible. Los tamaños relativamente cortos de los productos de la PCR, de aproximadamente 100-500 pb, generados con las pruebas de STR suelen ser compatibles con el ADN degradado que puede estar presente debido a las agresiones ambientales en el material biológico probatorio encontrado en la escena del crimen. La amplificación por PCR de varios loci de STR simultáneamente, o multiplexación, es posible con tintes fluorescentes de diferentes colores y productos de PCR de diferentes tamaños. El uso de múltiples loci permite un alto poder de discriminación en una sola prueba sin consumir mucho ADN (por ejemplo, 1 ng o menos de material de partida). Cabe destacar que estos loci STR centrales se encuentran entre los genes en los que se tolera un alto grado de variabilidad y, por tanto, no son directamente responsables de rasgos físicos como el color del pelo o de los ojos o de enfermedades genéticas.
Kits comerciales
Los kits comerciales, que proporcionan cebadores premezclados y una mezcla maestra estándar que contiene la polimerasa, los tampones enzimáticos y los dNTP, simplifican la generación de perfiles de STR y proporcionan resultados sobre un conjunto uniforme de loci de STR básicos para hacer posible el intercambio nacional e internacional de perfiles de ADN criminal. En la mayoría de los laboratorios forenses se prefieren los kits comerciales a los ensayos internos, aunque sean más caros, ya que ayudan a simplificar y estandarizar los procedimientos y eliminan la carga del control de calidad de los componentes de la PCR del ocupado usuario final. Además, los kits de STR suministran escalas alélicas que contienen alelos de STR comunes que han sido previamente caracterizados para el número de unidades de repetición mediante la secuenciación del ADN. Estas escalas alélicas se utilizan para calibrar los tamaños de los productos de PCR con el número de repeticiones de STR con fines de genotipado. La Figura 1 muestra la escalera alélica del ampliamente utilizado kit AmpFlSTR Identifiler (Applied Biosystems, Foster City, CA, USA) (3) que contiene 205 alelos a través de 16 loci coamplificados -15 STRs- más un ensayo de tipificación del sexo de la amelogenina.
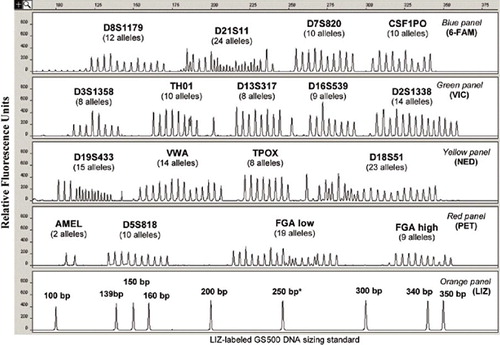
La determinación del genotipo en muestras procesadas posteriormente se realiza comparando el tamaño de los alelos (en relación con un estándar de tamaño interno) con una escalera alélica del kit STR suministrada comercialmente con números de repetición calibrados, cuyo tamaño se ajusta al mismo estándar de tamaño interno. Obsérvese que el pico de 250 pb en el estándar de tamaño GS500 no suele utilizarse debido a la migración anómala. Reimpreso con permiso de la Referencia 1, Figura 5.6.
El proceso completo de tipificación de STR incluye la recogida de muestras, la extracción de ADN, la cuantificación del ADN, la amplificación por PCR de múltiples loci de STR, la separación y el dimensionamiento de los alelos de STR, la tipificación de STR y la interpretación del perfil, y un informe de la importancia estadística de una coincidencia (si se observa). En muchas situaciones de trabajo de casos, como las pruebas de agresión sexual, las mezclas de ADN pueden ser el resultado de una combinación de fluidos corporales de la víctima y del agresor y crear un resultado complejo y difícil de interpretar.
Separaciones de ADN
Después de la amplificación por PCR, se mide la longitud total del amplicón de STR para determinar el número de repeticiones presentes en cada alelo encontrado en el perfil de ADN. Esta medición de la longitud se realiza a través de una separación basada en el tamaño que implica el gel o la electroforesis capilar (CE). Cada amplicón de STR ha sido marcado con fluorescencia durante la PCR, ya que el cebador específico del locus delantero o inverso contiene un tinte fluorescente. Así, registrando el color del tinte y el tiempo de migración de cada fragmento de ADN en relación con un estándar de tamaño interno, se puede determinar el tamaño de cada alelo de STR tras su separación de otros alelos de STR. Los instrumentos comúnmente utilizados para la separación y el dimensionamiento de alelos de STR incluyen los analizadores genéticos ABI PRISM 310 y ABI PRISM 3100 (Applied Biosystems) (4).
Hay una serie de artefactos tanto biológicos como instrumentales que a menudo deben ser clasificados para generar un perfil de STR completo y preciso (5; véase también la Referencia 1, Capítulos 6 y 15). Los artefactos biológicos incluyen productos de tartamudeo, picos divididos por adenilación incompleta, patrones trialélicos y alelos variantes que contienen mutaciones en las regiones de repetición o flanqueo que hacen que un alelo esté fuera de la escalera. Los artefactos instrumentales surgen de los picos de voltaje, las manchas de tinte y el sangrado entre los colores del tinte.
Aunque la instrumentación de CE de detección de fluorescencia multicolor, como el analizador genético ABI PRISM 3100, domina actualmente el campo, se están realizando esfuerzos para desarrollar plataformas de CE de microchip (6) para realizar separaciones de ADN de alta resolución con la eventual integración de la amplificación de PCR y las separaciones de CE (7). Además, se han utilizado técnicas de espectrometría de masas (EM) con desorción/ionización láser asistida por matriz (MALDI) y de ionización por electrospray (ESI) para la tipificación de STR sin escalas alélicas (8,9).
miniSTRs: Recuperación de la información del ADN degradado
Las moléculas de ADN que se exponen al agua y/o al calor, con el tiempo, comenzarán a descomponerse en trozos más pequeños. Varios estudios han demostrado lo que se predice teóricamente: que los tipos de ADN pueden recuperarse más eficazmente de las muestras de ADN degradado cuando los productos de PCR son más pequeños (10-12). Moviendo los cebadores de la PCR más cerca de la región de repetición del STR, los tamaños de los productos pueden reducirse conservando la misma información (11). La utilidad de los ensayos miniSTR se ha confirmado en estudios intra e interlaboratorios con muestras óseas degradadas y manchas de sangre y saliva envejecidas (12). Las tasas de éxito en la recuperación de información de muestras de ADN comprometidas mejoran con los sistemas miniSTR en comparación con los kits de STR convencionales. Se puede encontrar una línea de tiempo que cubre el desarrollo de los miniSTRs en www.cstl.nist.gov/biotech/strbase/miniSTR/timeline.htm.
Usando su química de 5 tintes y la tecnología de modificadores de movilidad, Applied Biosystems ha desarrollado un kit de miniSTR capaz de amplificar ocho loci de STR básicos y amelogenina con tamaños de producto de PCR reducidos en relación con los kits comerciales actuales. Este kit AmpFlSTR MiniFiler, que incluye una mezcla maestra de PCR mejorada, debería ayudar a recuperar los resultados de las muestras de ADN degradadas. Sin embargo, es importante tener en cuenta que debido a que se utilizan diferentes cebadores de PCR con el kit miniSTR en relación con los kits de STR anteriores, pueden producirse resultados discordantes debido a las mutaciones del sitio de unión del cebador que causan la caída del alelo.
Prueba de concordancia
Algunos alelos de STR contienen variación de la secuencia en las regiones flanqueantes. Si se produce un cambio de nucleótidos (o una inserción o supresión) en un sitio de unión del cebador de PCR en un alelo particular, es posible que la secuencia mutante sea incompatible con el recocido del cebador y que el alelo variante no se amplifique (1). Este abandono alélico se denomina a veces alelo nulo. Una muestra que en realidad es un heterocigoto puede aparecer como un aparente homocigoto si los cebadores de la PCR no consiguen anillarse y amplificar el alelo que contiene la mutación puntual (Figura 2). En algunos casos, el alelo nulo puede recuperarse reduciendo la temperatura de recocido, lo que permite una unión menos estricta entre el cebador y la plantilla de ADN. Un estudio de concordancia, en el que se analiza un conjunto de las mismas muestras de ADN con cebadores de PCR que no se solapan, permite la detección de alelos nulos. Un examen de MiniFiler frente a Identifiler sólo encontró 27 discrepancias en más de 10.000 genotipos comparados (13). Un resumen de los resultados discordantes observados en los estudios de concordancia está disponible en www.cstl.nist.gov/biotech/strbase/NullAlleles.htm.
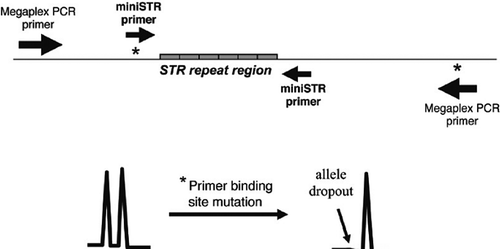
Generalmente, las regiones de flanqueo alrededor de las repeticiones de STR son consistentes entre varios alelos, permitiendo una amplificación robusta del locus STR. Los asteriscos representan posibles mutaciones en los sitios de unión de los cebadores que podrían conducir a la pérdida de alelos y a la discordancia entre los conjuntos de cebadores probados (panel inferior).
Si se utilizan los mismos cebadores de PCR, se pueden esperar resultados idénticos de tipificación de STR en la misma muestra de ADN. Sin embargo, si se cambian las posiciones de los cebadores, existe la posibilidad de que se produzcan abandonos de alelos o cambios de tamaño. En algunos casos, los cebadores adicionales que coinciden con el alelo mutante -a menudo denominados cebadores degenerados- pueden incluirse en la mezcla de cebadores del kit para permitir la recuperación de la amplificación de la PCR cuando el alelo variante que contiene un desajuste en el sitio de unión del cebador está presente (14).
Perspectivas sobre el futuro
Como señaló hace casi 7 años el Grupo de Trabajo de Investigación y Desarrollo de la Comisión Nacional sobre el Futuro de las Pruebas de ADN (15), los STRs serán probablemente los marcadores de elección en el futuro inmediato debido a su uso generalizado en las bases de datos nacionales de ADN (2,16,17). Es probable que veamos una progresión evolutiva en la tecnología de tipificación, métodos de amplificación más rápidos, kits de STR adicionales que contengan nuevos loci y programas informáticos que permitan una evaluación rápida de los datos de tipificación de STR. Gracias a los continuos avances, las tecnologías de tipificación de STR podrán miniaturizarse e integrarse con otras partes del proceso, como la extracción y la amplificación del ADN. En la actualidad se están construyendo bases de datos de ADN eficaces y se están resolviendo numerosos casos forenses mediante la generación de perfiles de STR con un conjunto común de marcadores genéticos. Estos loci centrales actuales han desempeñado y seguirán desempeñando un papel vital en las pruebas de identidad humana.
Agradecimientos
El autor, John M. Butler, está financiado por el Instituto Nacional de Justicia a través del acuerdo interinstitucional 2003-IJ-R-029 con la Oficina de Estándares de Aplicación de la Ley del Instituto Nacional de Estándares y Tecnología (NIST). Los puntos de vista que aparecen en este documento son los del autor y no representan necesariamente la posición o las políticas oficiales del Departamento de Justicia de los Estados Unidos. Algunos equipos, instrumentos y materiales comerciales se identifican para especificar los procedimientos experimentales de la forma más completa posible. En ningún caso dicha identificación implica una recomendación o respaldo por parte del NIST ni implica que cualquiera de los materiales, instrumentos o equipos identificados sean necesariamente los mejores disponibles para el propósito. Los comentarios sobre este manuscrito de Peter Vallone (Equipo del Proyecto de Identidad Humana del NIST) fueron muy apreciados.
Declaración de intereses en competencia
El autor declara no tener intereses en competencia.
- 1. Butler, J.M. 2005. Forensic DNA Typing: Biology, Technology, and Genetics of STR Markers, 2nd ed. Elsevier Academic Press, New York.Google Scholar
- 2. Butler, J.M. 2006. Genetics and genomics of core STR loci used in human identity testing. J. Forensic Sci. 51:253-265.Crossref, Medline, CAS, Google Scholar
- 3. Collins, P.J., L.K. Hennessy, C.S. Leibelt, R.K. Roby, D.J. Reeder y P.A. Foxall. 2004. Developmental validation of a singletube amplification of the 13 CODIS STR loci, D2S1338, D19S433, and amelogenin: the AmpFlSTR Identifiler PCR Amplification Kit. J. Forensic Sci. 49:1265-1277.Crossref, Medline, CAS, Google Scholar
- 4. Butler, J.M., E. Buel, F. Crivellente, y B.R. McCord. 2004. Tipificación forense del ADN mediante electroforesis capilar: uso de los analizadores genéticos ABI Prism 310 y 3100 para el análisis de STR. Electrophoresis 25:1397-1412.Crossref, Medline, CAS, Google Scholar
- 5. Grupo de trabajo científico sobre métodos de análisis de ADN (SWGDAM). 2000. Directrices de interpretación de repeticiones cortas en tándem (STR). Forensic Sci. Comm. 2(3): en línea en www.fbi.gov/hq/lab/fsc/backissu/july2000/strig.htm.Google Scholar
- 6. Yeung, S.H., S.A. Greenspoon, A. McGuckian, C.A. Crouse, C.A. Emrich, J. Ban y R.A. Mathies. 2006. Rapid and high-throughput forensic short tandem repeat typing using a 96-lane microfabricated capillary array electrophoresis microdevice. J. Forensic Sci. 51:740-747.Crossref, Medline, CAS, Google Scholar
- 7. Liu, P., T.S. Seo, N. Beyor, K.J. Shin, J.R. Scherer, and R.A. Mathies. 2007. Integrated portable polymerase chain reaction-capillary electrophoresis microsystem for rapid forensic short tandem repeat typing. Anal. Chem. 79:1881-1889.Crossref, Medline, CAS, Google Scholar
- 8. Butler, J.M., J. Li, T.A. Shaler, J.A. Monforte, and C.H. Becker. 1998. Reliable genotyping of short tandem repeat loci without an allelic ladder using time-of-flight mass spectrometry. Int. J. Legal Med. 112:45-49.Crossref, Medline, CAS, Google Scholar
- 9. Oberacher, H., F. Pitterl, G. Huber, H. Niederstatter, M. Steinlechner y W. Parson. En prensa. Incremento de la eficacia forense de las huellas de ADN mediante la resolución simultánea de la variabilidad de longitudes y nucleótidos por espectrometría de masas de alto rendimiento. Hum. Mutat. Google Scholar
- 10. Wiegand, P. y M. Kleiber. 2001. Less is more-length reduction of STR amplicons using redigned primers. Int. J. Legal Med. 114:285-287.Crossref, Medline, CAS, Google Scholar
- 11. Butler, J.M., Y. Shen, y B.R. McCord. 2003. The development of reduced size STR amplicons as tools for analysis of degraded DNA. J. Forensic Sci. 48:1054-1064.Crossref, Medline, CAS, Google Scholar
- 12. Dixon, L.A., A.E. Dobbins, H.K. Pulker, J.M. Butler, P.M. Vallone, M.D. Coble, W. Parson, B. Berger, et al.. 2006. Analysis of artificially degraded DNA using STRs and SNPs-results of a collaborative European (EDNAP) exercise. Forensic Sci. Int. 164:33-44.Crossref, Medline, CAS, Google Scholar
- 13. Hill, C.R., M.C. Kline, J.J. Mulero, R.E. Lagace, C.W. Chang, L.K. Hennessy y J.M. Butler. 2007. Estudio de concordancia entre el kit de amplificación PCR AmpFlSTR MiniFiler y los kits convencionales de tipificación de STR. J. Forensic Sci. 52:870-873.Crossref, Medline, CAS, Google Scholar
- 14. Leibelt, C., B. Budowle, P. Collins, Y. Daoudi, T. Moretti, G. Nunn, D. Reeder y R. Roby. 2003. Identification of a D8S1179 primer binding site mutation and the validation of a primer designed to recover null alleles. Forensic Sci. Int. 133:220-227.Crossref, Medline, CAS, Google Scholar
- 15. Instituto Nacional de Justicia (NIJ). 2000. The future of forensic DNA testing: predictions of the Research and Development Working Group of the National Commission on the Future of DNA Evidence, Washington, D.C. www.ojp.usdoj.gov/nij/pubs-sum/183697.htm.Google Scholar
- 16. Gill, P. 2002. Role of short tandem repeat DNA in forensic casework in the UK-past, present, and future perspectives. BioTechniques 32:366-372.Link, CAS, Google Scholar
- 17. Gill, P., D.J. Werrett, B. Budowle, y R. Guerrieri. 2004. An assessment of whether SNPs will replace STRs in national DNA databases-joint considerations of the DNA working group of the European Network of Forensic Science Institutes (ENFSI) and the Scientific Working Group on DNA Analysis Methods (SWGDAM). Sci. Justice 44:51-53.Crossref, Medline, Google Scholar