Wprowadzenie
Krótkie powtórzenia tandemowe (STRs), które są czasami określane jako mikrosatelity lub powtórzenia sekwencji prostej (SSRs), są akordeonowymi odcinkami DNA zawierającymi rdzeniowe jednostki powtórzenia o długości od dwóch do siedmiu nukleotydów, które powtarzają się tandemowo od około pół tuzina do kilkudziesięciu razy (1). Chociaż ludzki genom zawiera tysiące na tysiące markerów STR, tylko niewielki podstawowy zestaw loci został wybrany do wykorzystania w kryminalistycznych badaniach DNA i tożsamości ludzi (2). Podobnie jak używanie jednej, wspólnej waluty w sensie finansowym, podstawowe loci pozwalają na wymianę i porównywanie równoważnych informacji genetycznych. Obecnie dostępne są komercyjne zestawy do generowania profili DNA zawierających te podstawowe loci STR (Tabela 1). Miliony profili STR są generowane każdego roku na całym świecie przez laboratoria rządowe, uniwersyteckie i prywatne, wykonujące różne formy badania tożsamości ludzi, w tym bazy danych DNA, kryminalistykę sądową, identyfikację osób zaginionych/ofiar katastrof masowych lub badanie ojcostwa.
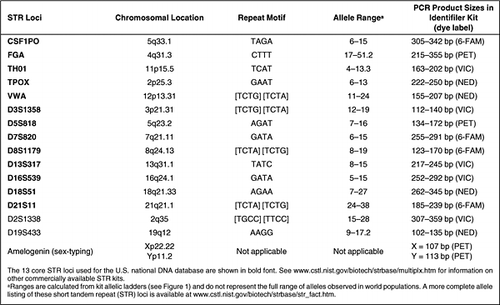
W przypadku typowania STR, PCR jest wykorzystywany do odzyskiwania informacji z niewielkich ilości dostępnego materiału biologicznego. Stosunkowo krótkie rozmiary produktów PCR, wynoszące około 100-500 bp, generowane podczas badania STR są generalnie zgodne ze zdegradowanym DNA, które może być obecne w wyniku działania czynników środowiskowych na dowodowym materiale biologicznym znalezionym na miejscu przestępstwa. Amplifikacja PCR wielu loci STR jednocześnie, lub multipleksowanie, jest możliwe przy użyciu różnokolorowych barwników fluorescencyjnych i produktów PCR o różnej wielkości. Użycie wielu loci umożliwia wysoką moc dyskryminacji w pojedynczym teście bez zużywania dużej ilości DNA (np. 1 ng lub mniej materiału wyjściowego). Warto zauważyć, że te podstawowe loci STR występują pomiędzy genami, w których tolerowany jest wysoki stopień zmienności, a zatem nie są bezpośrednio odpowiedzialne za cechy fizyczne, takie jak kolor włosów lub oczu, czy też choroby genetyczne.
Komercyjne zestawy
Komercyjne dostępne zestawy, które dostarczają wstępnie zmieszane primery i standardową mieszaninę wzorcową zawierającą polimerazę, bufory enzymatyczne i dNTP, upraszczają generowanie profili STR i dostarczają wyniki na jednolitym zestawie rdzeniowych loci STR, aby umożliwić krajowe i międzynarodowe dzielenie się kryminalnymi profilami DNA. Komercyjne zestawy są preferowane w większości laboratoriów kryminalistycznych w porównaniu z testami wewnętrznymi, nawet jeśli są one droższe, ponieważ zestawy te pomagają uprościć i ustandaryzować procedury oraz zdejmują ciężar kontroli jakości składników PCR z zapracowanego użytkownika końcowego. Ponadto, zestawy STR dostarczają drabinki alleliczne zawierające powszechne allele STR, które zostały wcześniej scharakteryzowane pod względem liczby jednostek powtórzenia poprzez sekwencjonowanie DNA. Te drabinki alleliczne są używane do kalibracji wielkości produktów PCR do liczby powtórzeń STR dla celów genotypowania. Rysunek 1 przedstawia drabinkę alleliczną dla szeroko stosowanego zestawu AmpFlSTR Identifiler (Applied Biosystems, Foster City, CA, USA) (3) zawierającą 205 alleli w 16 koamplifikowanych loci – 15 STR plus test typowania płci amelogeniny.
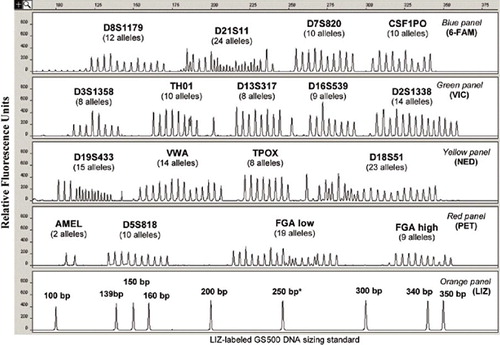
Określenie genotypu w później przetworzonych próbkach jest wykonywane przez porównanie rozmiaru alleli (w stosunku do wewnętrznego standardu rozmiaru) z komercyjnie dostarczoną drabinką alleliczną zestawu STR ze skalibrowanymi numerami powtórzeń, która jest wymiarowana zgodnie z tym samym wewnętrznym standardem rozmiaru. Należy zauważyć, że pik 250-bp w standardzie rozmiaru GS500 nie jest zwykle używany z powodu anomalnej migracji. Przedrukowano za pozwoleniem z odnośnika 1, Rysunek 5.6.
Pełny proces typowania STR obejmuje pobieranie próbek, ekstrakcję DNA, kwantyfikację DNA, amplifikację PCR wielu loci STR, rozdzielanie alleli STR i określanie wielkości, typowanie STR i interpretację profilu oraz raport o statystycznej istotności zgodności (jeśli zaobserwowano). W wielu przypadkach, takich jak dowody napaści na tle seksualnym, mieszaniny DNA mogą wynikać z połączenia płynów ustrojowych ofiary i sprawcy, tworząc złożone i trudne do interpretacji wyniki.
Rozdzielanie DNA
Po amplifikacji PCR, całkowita długość amplikonu STR jest mierzona w celu określenia liczby powtórzeń obecnych w każdym allelu znalezionym w profilu DNA. Ten pomiar długości jest wykonywany poprzez separację opartą na wielkości z wykorzystaniem żelu lub elektroforezy kapilarnej (CE). Każdy amplikon STR został wyznakowany fluorescencyjnie podczas PCR, ponieważ primer forward lub reverse locus-specific zawiera barwnik fluorescencyjny. Tak więc, rejestrując kolor barwnika i czas migracji każdego fragmentu DNA w stosunku do wewnętrznego standardu wielkości, można określić wielkość każdego allelu STR po jego oddzieleniu od innych alleli STR. Powszechnie stosowane instrumenty do rozdzielania alleli STR i określania ich wielkości obejmują analizatory genetyczne ABI PRISM 310 i ABI PRISM 3100 (Applied Biosystems) (4).
Istnieje szereg artefaktów zarówno biologicznych, jak i instrumentalnych, które często muszą być posortowane w celu wygenerowania kompletnego i dokładnego profilu STR (5; patrz również odnośnik 1, rozdziały 6 i 15). Artefakty biologiczne obejmują produkty jąkania, rozszczepione piki z niepełnej adenylacji, wzory trialleliczne oraz warianty alleli zawierające mutacje w powtórzeniu lub regionach flankujących, które powodują, że allel jest poza linią. Artefakty instrumentalne powstają w wyniku skoków napięcia, plam barwnika i przebić pomiędzy kolorami barwnika.
Pomimo, że wielokolorowe instrumenty CE z detekcją fluorescencji, takie jak analizator genetyczny ABI PRISM 3100, obecnie dominują w tej dziedzinie, trwają wysiłki w celu opracowania mikrochipowych platform CE (6) do wykonywania rozdzielania DNA o wysokiej rozdzielczości z ewentualną integracją amplifikacji PCR i rozdzielania CE (7). Ponadto, spektrometria mas (MS) z technikami desorpcji/jonizacji laserowej wspomaganej matrycą (MALDI) i jonizacji elektrospray (ESI) została wykorzystana do typowania STR bez drabinek allelicznych (8,9).
miniSTRs: Recovery of Information From Degraded DNA
Cząsteczki DNA, które są wystawione na działanie wody i/lub ciepła, z czasem zaczną się rozpadać na mniejsze kawałki. Wiele badań wykazało to, co jest teoretycznie przewidywane – że typy DNA mogą być odzyskiwane bardziej efektywnie ze zdegradowanych próbek DNA, gdy produkty PCR są mniejsze (10-12). Poprzez przesunięcie primerów PCR bliżej regionu powtórzenia STR, rozmiary produktów mogą być zmniejszone przy zachowaniu tej samej informacji (11). Użyteczność testów miniSTR została potwierdzona w badaniach wewnątrz- i międzylaboratoryjnych obejmujących zdegradowane próbki kości oraz plamy krwi i śliny (12). Wskaźniki sukcesu w odzyskiwaniu informacji z uszkodzonych próbek DNA są lepsze w przypadku systemów miniSTR w porównaniu z konwencjonalnymi zestawami STR. Oś czasu obejmującą rozwój miniSTR można znaleźć na stronie www.cstl.nist.gov/biotech/strbase/miniSTR/timeline.htm.
Przy użyciu chemii 5-dye i technologii modyfikatorów ruchliwości firma Applied Biosystems opracowała zestaw miniSTR zdolny do amplifikacji ośmiu podstawowych loci STR i amelogeniny przy zmniejszonych rozmiarach produktów PCR w porównaniu z obecnymi zestawami komercyjnymi. Ten zestaw AmpFlSTR MiniFiler, który zawiera ulepszoną mieszaninę wzorcową PCR, powinien wspomóc wysiłki zmierzające do odzyskania wyników ze zdegradowanych próbek DNA. Należy jednak pamiętać, że ponieważ w zestawie miniSTR stosowane są inne startery PCR niż w poprzednich zestawach STR, mogą wystąpić rozbieżne wyniki z powodu mutacji w miejscu wiązania startera, które powodują zanikanie alleli.
Testowanie zgodności
Niektóre allele STR zawierają zmienność sekwencji w regionach flankujących. Jeżeli zmiana nukleotydów (lub insercja lub delecja) występuje w miejscu wiązania startera PCR w danym allelu, wówczas możliwe jest, że zmutowana sekwencja jest niezgodna z annealingiem startera i allel wariantu nie amplifikuje się (1). Ten zanik allelu jest czasami określany jako allel zerowy. Próbka, która jest w rzeczywistości heterozygotą może pojawić się jako pozorna homozygota, jeżeli startery PCR nie zdołają się zespolić i amplifikować allelu zawierającego mutację punktową (Rysunek 2). W niektórych przypadkach, allel zerowy może zostać odzyskany poprzez obniżenie temperatury annealingu, pozwalając na mniej rygorystyczne wiązanie pomiędzy starterem a szablonem DNA. Badanie zgodności, w którym testuje się zestaw tych samych próbek DNA z nienakładającymi się primerami PCR, pozwala na wykrycie alleli zerowych. Badanie MiniFiler w porównaniu z Identifiler wykazało tylko 27 rozbieżności w ponad 10 000 porównywanych genotypów (13). Podsumowanie niezgodnych wyników zaobserwowanych w badaniach zgodności jest dostępne na stronie www.cstl.nist.gov/biotech/strbase/NullAlleles.htm.
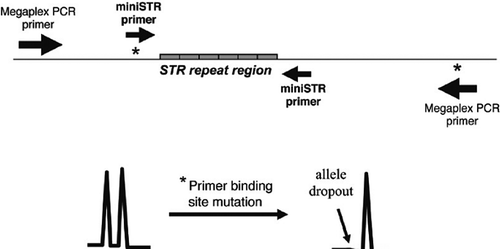
Ogólnie, regiony flankujące wokół powtórzeń STR są spójne pomiędzy różnymi allelami, umożliwiając silną amplifikację locus STR. Gwiazdki reprezentują potencjalne mutacje w miejscach wiązania primerów, które mogą prowadzić do wypadania alleli i niezgodności pomiędzy testowanymi zestawami primerów (panel dolny).
Dopóki używane są te same primery PCR, można oczekiwać identycznych wyników typowania STR na tej samej próbce DNA. Jednakże, jeśli pozycje primerów są zmienione, istnieje możliwość, że allele zostaną odrzucone lub nastąpi przesunięcie rozmiaru. W niektórych przypadkach, dodatkowe startery odpowiadające zmutowanemu allelowi – często określane jako startery zdegenerowane – mogą być włączone do mieszaniny starterów zestawu, aby umożliwić odzyskanie amplifikacji PCR, gdy obecny jest allel wariantu zawierający niedopasowanie w miejscu wiązania startera (14).
Perspektywy na przyszłość
Jak zauważono prawie 7 lat temu przez Grupę Roboczą ds. Badań i Rozwoju Narodowej Komisji ds. Przyszłości Dowodów DNA (15), STRs będą prawdopodobnie markerami z wyboru w przewidywalnej przyszłości z powodu ich powszechnego stosowania w krajowych bazach danych DNA (2,16,17). Prawdopodobnie będziemy świadkami ewolucyjnego postępu w technologii typowania, szybszych metod amplifikacji, dodatkowych zestawów STR zawierających nowe loci oraz programów komputerowych, które umożliwią szybką ocenę danych typowania STR. Dzięki ciągłemu postępowi, technologie typowania STR mogą zostać zminiaturyzowane i zintegrowane z innymi częściami procesu, takimi jak ekstrakcja i amplifikacja DNA. Tworzone są skuteczne bazy danych DNA, a liczne sprawy kryminalistyczne są rozwiązywane już dzisiaj poprzez generowanie profili STR ze wspólnym zestawem markerów genetycznych. Te obecne podstawowe loci odgrywały i nadal będą odgrywać istotną rolę w badaniu tożsamości ludzi.
Podziękowania
Autor, John M. Butler, jest finansowany przez Narodowy Instytut Sprawiedliwości poprzez umowę międzyagencyjną 2003-IJ-R-029 z Narodowym Instytutem Norm i Technologii (NIST) Biuro Norm Egzekwowania Prawa. Poglądy przedstawione w tym dokumencie są poglądami autora i nie muszą reprezentować oficjalnego stanowiska lub polityki Departamentu Sprawiedliwości USA. Niektóre komercyjne urządzenia, instrumenty i materiały zostały zidentyfikowane w celu jak najdokładniejszego określenia procedur eksperymentalnych. W żadnym wypadku taka identyfikacja nie oznacza rekomendacji lub poparcia ze strony NIST, ani też nie oznacza, że jakiekolwiek materiały, przyrządy lub sprzęt są koniecznie najlepsze z dostępnych do danego celu. Uwagi dotyczące tego manuskryptu autorstwa Petera Vallone (NIST Human Identity Project Team) zostały bardzo docenione.
Oświadczenie o interesach konkurencyjnych
Autor nie zgłasza żadnych interesów konkurencyjnych.
- 1. Butler, J.M. 2005. Forensic DNA Typing: Biology, Technology, and Genetics of STR Markers, 2nd ed. Elsevier Academic Press, New York.Google Scholar
- 2. Butler, J.M. 2006. Genetyka i genomika podstawowych loci STR używanych w badaniu tożsamości człowieka. J. Forensic Sci. 51:253-265.Crossref, Medline, CAS, Google Scholar
- 3. Collins, P.J., L.K. Hennessy, C.S. Leibelt, R.K. Roby, D.J. Reeder, and P.A. Foxall. 2004. Developmental validation of a singletube amplification of the 13 CODIS STR loci, D2S1338, D19S433, and amelogenin: the AmpFlSTR Identifiler PCR Amplification Kit. J. Forensic Sci. 49:1265-1277.Crossref, Medline, CAS, Google Scholar
- 4. Butler, J.M., E. Buel, F. Crivellente, and B.R. McCord. 2004. Forensic DNA typing by capillary electrophoresis: using the ABI Prism 310 and 3100 Genetic Analyzers for STR analysis. Electrophoresis 25:1397-1412.Crossref, Medline, CAS, Google Scholar
- 5. Naukowa Grupa Robocza ds. Metod Analizy DNA (SWGDAM). 2000. Wytyczne interpretacji krótkich powtórzeń tandemowych (STR). Forensic Sci. Comm. 2(3): online na www.fbi.gov/hq/lab/fsc/backissu/july2000/strig.htm.Google Scholar
- 6. Yeung, S.H., S.A. Greenspoon, A. McGuckian, C.A. Crouse, C.A. Emrich, J. Ban, and R.A. Mathies. 2006. Rapid and high-throughput forensic short tandem repeat typing using a 96-lane microfabricated capillary array electrophoresis microdevice. J. Forensic Sci. 51:740-747.Crossref, Medline, CAS, Google Scholar
- 7. Liu, P., T.S. Seo, N. Beyor, K.J. Shin, J.R. Scherer, and R.A. Mathies. 2007. Integrated portable polymerase chain reaction-capillary electrophoresis microsystem for rapid forensic short tandem repeat typing. Anal. Chem. 79:1881-1889.Crossref, Medline, CAS, Google Scholar
- 8. Butler, J.M., J. Li, T.A. Shaler, J.A. Monforte, and C.H. Becker. 1998. Wiarygodne genotypowanie loci krótkich powtórzeń tandemowych bez drabiny allelicznej przy użyciu spektrometrii masowej czasu przelotu. Int. J. Legal Med. 112:45-49.Crossref, Medline, CAS, Google Scholar
- 9. Oberacher, H., F. Pitterl, G. Huber, H. Niederstatter, M. Steinlechner i W. Parson. In press. Increased forensic efficiency of DNA fingerprints through simultaneous resolution of length and nucleotide variability by high-performance mass spectrometry. Hum. Mutat. Google Scholar
- 10. Wiegand, P. i M. Kleiber. 2001. Less is more – redukcja długości amplikonów STR przy użyciu przeprojektowanych starterów. Int. J. Legal Med. 114:285-287.Crossref, Medline, CAS, Google Scholar
- 11. Butler, J.M., Y. Shen, and B.R. McCord. 2003. Rozwój amplikonów STR o zmniejszonym rozmiarze jako narzędzi do analizy zdegradowanego DNA. J. Forensic Sci. 48:1054-1064.Crossref, Medline, CAS, Google Scholar
- 12. Dixon, L.A., A.E. Dobbins, H.K. Pulker, J.M. Butler, P.M. Vallone, M.D. Coble, W. Parson, B. Berger, et al. 2006. Analysis of artificially degraded DNA using STRs and SNPs-results of a collaborative European (EDNAP) exercise. Forensic Sci. Int. 164:33-44.Crossref, Medline, CAS, Google Scholar
- 13. Hill, C.R., M.C. Kline, J.J. Mulero, R.E. Lagace, C.W. Chang, L.K. Hennessy, and J.M. Butler. 2007. Concordance study between the AmpFlSTR MiniFiler PCR amplification kit and conventional STR typing kits. J. Forensic Sci. 52:870-873.Crossref, Medline, CAS, Google Scholar
- 14. Leibelt, C., B. Budowle, P. Collins, Y. Daoudi, T. Moretti, G. Nunn, D. Reeder, and R. Roby. 2003. Identification of a D8S1179 primer binding site mutation and the validation of a primer designed to recover null alleles. Forensic Sci. Int. 133:220-227.Crossref, Medline, CAS, Google Scholar
- 15. National Institute of Justice (NIJ). 2000. The future of forensic DNA testing: predictions of the Research and Development Working Group of the National Commission on the Future of DNA Evidence, Washington, D.C. www.ojp.usdoj.gov/nij/pubs-sum/183697.htm.Google Scholar
- 16. Gill, P. 2002. Rola krótkich tandemowych powtórzeń DNA w sprawach sądowych w Wielkiej Brytanii – przeszłość, teraźniejszość i perspektywy na przyszłość. BioTechniques 32:366-372.Link, CAS, Google Scholar
- 17. Gill, P., D.J. Werrett, B. Budowle, and R. Guerrieri. 2004. An assessment of whether SNPs will replace STRs in national DNA databases-joint considerations of the DNA working group of the European Network of Forensic Science Institutes (ENFSI) and the Scientific Working Group on DNA Analysis Methods (SWGDAM). Sci. Justice 44:51-53.Crossref, Medline, Google Scholar