Introduzione
Le ripetizioni in tandem brevi (STR), a volte definite microsatelliti o semplici ripetizioni di sequenza (SSR), sono tratti di DNA a forma di fisarmonica contenenti unità di ripetizione centrale di lunghezza compresa tra due e sette nucleotidi che si ripetono in tandem da circa una mezza dozzina a diverse decine di volte (1). Sebbene il genoma umano contenga migliaia e migliaia di marcatori STR, solo un piccolo nucleo di loci è stato selezionato per l’uso nel DNA forense e nei test di identità umana (2). Come l’uso di una moneta unica e comune in senso finanziario, i loci principali permettono di condividere e confrontare informazioni genetiche equivalenti. Sono ora disponibili kit commerciali per generare profili di DNA contenenti questi loci STR principali (Tabella 1). Milioni di profili STR vengono generati ogni anno in tutto il mondo da laboratori governativi, universitari e privati che eseguono varie forme di test dell’identità umana, tra cui la ricerca di dati sul DNA, i casi forensi, l’identificazione di persone scomparse/vittime di disastri di massa o i test di parentela.
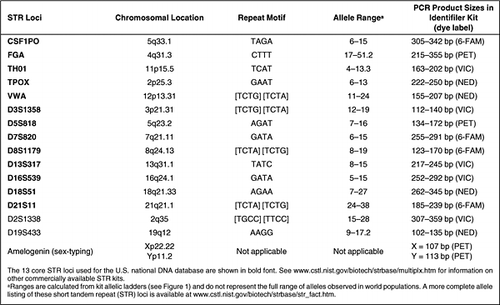
Con la tipizzazione STR, la PCR viene utilizzata per recuperare informazioni da piccole quantità di materiale biologico disponibile. Le dimensioni relativamente corte del prodotto PCR di circa 100-500 bp generato con il test STR sono generalmente compatibili con il DNA degradato che può essere presente a causa di insulti ambientali sul materiale biologico probatorio trovato sulla scena del crimine. L’amplificazione PCR di più loci STR contemporaneamente, o multiplexing, è possibile con coloranti fluorescenti di colore diverso e prodotti PCR di dimensioni diverse. L’uso di più loci permette un alto potere di discriminazione in un singolo test senza consumare molto DNA (ad esempio, 1 ng o meno di materiale di partenza). Vale la pena notare che questi loci STR si trovano tra i geni in cui è tollerato un alto grado di variabilità e non sono quindi direttamente responsabili di tratti fisici come il colore dei capelli o degli occhi o di malattie genetiche.
Kit commerciali
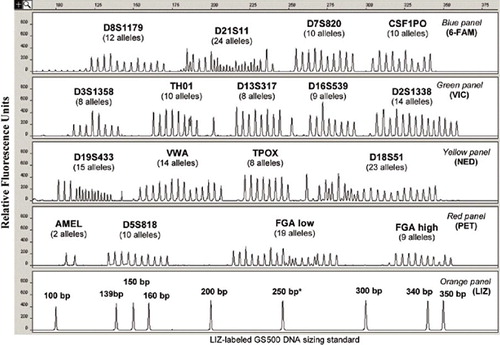
I kit disponibili in commercio, che forniscono primer premiscelati e una miscela master standard contenente la polimerasi, i tamponi enzimatici e i dNTP, semplificano la generazione di profili STR e forniscono risultati su un set uniforme di loci STR principali per rendere possibile la condivisione nazionale e internazionale dei profili di DNA criminale. I kit commerciali sono preferiti nella maggior parte dei laboratori forensi rispetto ai saggi interni, anche se i kit sono più costosi, poiché questi kit aiutano a semplificare e standardizzare le procedure e tolgono il peso del controllo di qualità dei componenti della PCR all’utente finale impegnato. Inoltre, i kit STR forniscono scale alleliche contenenti alleli STR comuni che sono stati precedentemente caratterizzati per il numero di unità di ripetizione tramite sequenziamento del DNA. Queste scale alleliche sono utilizzate per calibrare le dimensioni dei prodotti PCR al numero di ripetizioni STR ai fini della genotipizzazione. La Figura 1 mostra la scala allelica per il kit AmpFlSTR Identifiler ampiamente utilizzato (Applied Biosystems, Foster City, CA, USA) (3) che contiene 205 alleli su 16 loci coamplificati – 15 STR più un test di tipizzazione sessuale dell’amelogenina.
Il processo completo per la tipizzazione STR include la raccolta del campione, l’estrazione del DNA, la quantificazione del DNA, l’amplificazione PCR di più loci STR, la separazione e il dimensionamento degli alleli STR, la tipizzazione STR e l’interpretazione del profilo e un rapporto sulla significatività statistica di una corrispondenza (se osservata). In molte situazioni, come nel caso di prove di aggressioni sessuali, le miscele di DNA possono derivare da una combinazione di fluidi corporei della vittima e dell’aggressore e creare un risultato complesso e difficile da interpretare.
DNA Separations
Dopo l’amplificazione PCR, viene misurata la lunghezza complessiva dell’amplicon STR per determinare il numero di ripetizioni presenti in ciascun allele trovato nel profilo del DNA. Questa misurazione della lunghezza viene effettuata tramite una separazione basata sulle dimensioni che coinvolge il gel o l’elettroforesi capillare (CE). Ogni amplicone STR è stato marcato in modo fluorescente durante la PCR, poiché il primer specifico del locus forward o reverse contiene un colorante fluorescente. Pertanto, registrando il colore del colorante e il tempo di migrazione di ciascun frammento di DNA rispetto a uno standard di dimensione interna, è possibile determinare la dimensione di ciascun allele STR dopo la sua separazione da altri alleli STR. Gli strumenti comunemente usati per la separazione e il dimensionamento degli alleli STR includono gli analizzatori genetici ABI PRISM 310 e ABI PRISM 3100 (Applied Biosystems) (4).
Ci sono una serie di artefatti sia biologici che strumentali che spesso devono essere selezionati per generare un profilo STR completo e accurato (5; vedere anche la referenza 1, capitoli 6 e 15). Gli artefatti biologici includono prodotti balbuzienti, picchi divisi da adenilazione incompleta, modelli triallelici e alleli varianti contenenti mutazioni nella ripetizione o nelle regioni adiacenti che causano l’off-ladder di un allele. Gli artefatti strumentali derivano da picchi di tensione, blob di colorante e bleed-through tra i colori del colorante.
Mentre la strumentazione CE di rilevamento della fluorescenza multicolore, come l’analizzatore genetico ABI PRISM 3100, attualmente domina il campo, sono in corso sforzi per sviluppare piattaforme CE su microchip (6) per eseguire separazioni di DNA ad alta risoluzione con eventuale integrazione dell’amplificazione PCR e separazioni CE (7). Inoltre, la spettrometria di massa (MS) con tecniche di desorbimento/ionizzazione laser assistita da matrice (MALDI) e ionizzazione elettrospray (ESI) sono state utilizzate per la tipizzazione STR senza scale alleliche (8,9).
miniSTRs: Recupero di informazioni dal DNA degradato
Le molecole di DNA che sono esposte all’acqua e/o al calore, col tempo, iniziano a rompersi in pezzi più piccoli. Un certo numero di studi ha dimostrato ciò che è teoricamente previsto – che i tipi di DNA possono essere recuperati più efficacemente da campioni di DNA degradato quando i prodotti PCR sono più piccoli (10-12). Spostando i primer di PCR più vicino alla regione di ripetizione STR, le dimensioni del prodotto possono essere ridotte mantenendo le stesse informazioni (11). L’utilità dei test miniSTR è stata confermata in studi intra- e interlaboratorio che hanno coinvolto campioni ossei degradati e macchie di sangue e saliva invecchiate (12). Le percentuali di successo nel recupero di informazioni da campioni di DNA compromessi migliorano con i sistemi miniSTR rispetto ai kit STR convenzionali. Una timeline che copre lo sviluppo delle miniSTR può essere trovata su www.cstl.nist.gov/biotech/strbase/miniSTR/timeline.htm.
Utilizzando la loro chimica a 5 coloranti e la tecnologia del modificatore di mobilità, Applied Biosystems ha sviluppato un kit miniSTR in grado di amplificare otto loci STR principali e l’amelogenina con dimensioni del prodotto PCR ridotte rispetto agli attuali kit commerciali. Questo kit AmpFlSTR MiniFiler, che include una miscela master PCR migliorata, dovrebbe aiutare gli sforzi per recuperare i risultati da campioni di DNA degradati. Tuttavia, è importante tenere presente che, poiché con il kit miniSTR vengono utilizzati primer PCR diversi rispetto ai precedenti kit STR, possono verificarsi risultati discordanti a causa di mutazioni del sito di legame del primer che causano il dropout dell’allele.
Test di concordanza
Alcuni alleli STR contengono variazioni di sequenza nelle regioni adiacenti. Se si verifica un cambiamento nucleotidico (o un’inserzione o una delezione) in un sito di legame del primer PCR in un particolare allele, è possibile che la sequenza mutante sia incompatibile con l’annealing del primer e che l’allele variante non si amplifichi (1). Questo abbandono allelico è talvolta indicato come allele nullo. Un campione che in realtà è un eterozigote potrebbe apparire come un omozigote apparente se i primer della PCR non riescono ad annealing e ad amplificare l’allele contenente la mutazione puntiforme (Figura 2). In alcuni casi, l’allele nullo può essere recuperato abbassando la temperatura di annealing, permettendo un legame meno stretto tra il primer e il modello di DNA. Uno studio di concordanza, che analizza una serie di campioni dello stesso DNA con primer PCR non sovrapposti, permette di rilevare gli alleli nulli. Un esame di MiniFiler rispetto a Identifiler ha trovato solo 27 discrepanze su oltre 10.000 genotipi confrontati (13). Un riassunto dei risultati discordanti osservati negli studi di concordanza è disponibile su www.cstl.nist.gov/biotech/strbase/NullAlleles.htm.
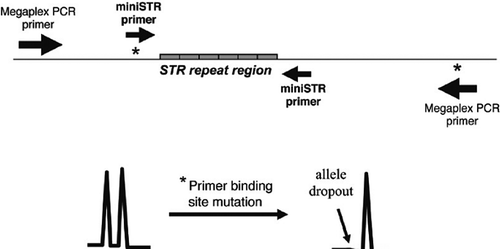
Generalmente, le regioni di accompagnamento intorno alle ripetizioni STR sono coerenti tra i vari alleli, consentendo una robusta amplificazione del locus STR. Gli asterischi rappresentano potenziali mutazioni del sito di legame del primer che potrebbero portare al dropout allelico e alla discordanza tra i set di primer testati (pannello inferiore).
Finché vengono utilizzati gli stessi primer PCR, ci si possono aspettare risultati di tipizzazione STR identici sullo stesso campione di DNA. Tuttavia, se le posizioni dei primer vengono cambiate, esiste la possibilità che si verifichi un dropout allelico o un size shifting. In alcuni casi, i primer extra che corrispondono all’allele mutante – spesso indicati come primer degenerati – possono essere inclusi nella miscela di primer del kit per consentire il recupero dell’amplificazione PCR quando è presente l’allele variante contenente un mismatch nel sito di legame del primer (14).
Prospettive sul futuro
Come notato quasi 7 anni fa dal gruppo di lavoro di ricerca e sviluppo della Commissione nazionale sul futuro delle prove del DNA (15), gli STR saranno probabilmente i marcatori di scelta per il prossimo futuro a causa del loro uso diffuso nei database nazionali del DNA (2,16,17). Probabilmente assisteremo a una progressione evolutiva della tecnologia di tipizzazione, a metodi di amplificazione più rapidi, a kit STR aggiuntivi contenenti nuovi loci e a programmi informatici che consentiranno una rapida valutazione dei dati di tipizzazione STR. Grazie ai continui progressi, le tecnologie di tipizzazione STR potrebbero diventare miniaturizzate e integrate con altre parti del processo, come l’estrazione e l’amplificazione del DNA. Si stanno costruendo efficaci database del DNA e numerosi casi forensi vengono risolti oggi attraverso la generazione di profili STR con un insieme comune di marcatori genetici. Questi attuali loci centrali hanno giocato e continueranno a giocare un ruolo vitale nei test di identità umana.
Riconoscimenti
L’autore, John M. Butler, è finanziato dal National Institute of Justice attraverso l’accordo interagenzia 2003-IJ-R-029 con il National Institute of Standards and Technology (NIST) Office of Law Enforcement Standards. I punti di vista in questo documento sono quelli dell’autore e non rappresentano necessariamente la posizione ufficiale o le politiche del Dipartimento di Giustizia degli Stati Uniti. Alcune attrezzature commerciali, strumenti e materiali sono identificati per specificare le procedure sperimentali nel modo più completo possibile. In nessun caso tale identificazione implica una raccomandazione o un’approvazione da parte del NIST né implica che uno qualsiasi dei materiali, strumenti o attrezzature identificati siano necessariamente i migliori disponibili per lo scopo. I commenti su questo manoscritto di Peter Vallone (NIST Human Identity Project Team) sono stati molto apprezzati.
Dichiarazione di interessi concorrenti
L’autore non dichiara interessi concorrenti.
- 1. Butler, J.M. 2005. Tipizzazione forense del DNA: Biology, Technology, and Genetics of STR Markers, 2nd ed. Elsevier Academic Press, New York.Google Scholar
- 2. Butler, J.M. 2006. Genetica e genomica dei loci STR principali utilizzati nei test di identità umana. J. Forensic Sci. 51:253-265.Crossref, Medline, CAS, Google Scholar
- 3. Collins, P.J., L.K. Hennessy, C.S. Leibelt, R.K. Roby, D.J. Reeder, and P.A. Foxall. 2004. Convalida dello sviluppo di un’amplificazione a tubo singolo dei 13 loci CODIS STR, D2S1338, D19S433 e amelogenina: il kit di amplificazione PCR AmpFlSTR Identifiler. J. Forensic Sci. 49:1265-1277.Crossref, Medline, CAS, Google Scholar
- 4. Butler, J.M., E. Buel, F. Crivellente, and B.R. McCord. 2004. Tipizzazione forense del DNA mediante elettroforesi capillare: utilizzo degli analizzatori genetici ABI Prism 310 e 3100 per l’analisi STR. Electrophoresis 25:1397-1412.Crossref, Medline, CAS, Google Scholar
- 5. Gruppo di lavoro scientifico sui metodi di analisi del DNA (SWGDAM). 2000. Linee guida per l’interpretazione delle ripetizioni tandem brevi (STR). Forensic Sci. Comm. 2(3): online su www.fbi.gov/hq/lab/fsc/backissu/july2000/strig.htm.Google Scholar
- 6. Yeung, S.H., S.A. Greenspoon, A. McGuckian, C.A. Crouse, C.A. Emrich, J. Ban, and R.A. Mathies. 2006. Tipizzazione forense rapida e ad alta produttività delle ripetizioni tandem brevi utilizzando un microdispositivo di elettroforesi capillare microfabbricato a 96 corsie. J. Forensic Sci. 51:740-747.Crossref, Medline, CAS, Google Scholar
- 7. Liu, P., T.S. Seo, N. Beyor, K.J. Shin, J.R. Scherer, and R.A. Mathies. 2007. Microsistema portatile integrato di reazione a catena della polimerasi-elettroforesi capillare per una rapida tipizzazione forense delle ripetizioni tandem brevi. Anal. Chem. 79:1881-1889.Crossref, Medline, CAS, Google Scholar
- 8. Butler, J.M., J. Li, T.A. Shaler, J.A. Monforte, e C.H. Becker. 1998. Genotipizzazione affidabile di loci a ripetizione tandem corta senza una scala allelica utilizzando la spettrometria di massa time-of-flight. Int. J. Legal Med. 112:45-49.Crossref, Medline, CAS, Google Scholar
- 9. Oberacher, H., F. Pitterl, G. Huber, H. Niederstatter, M. Steinlechner, e W. Parson. In stampa. Maggiore efficienza forense di impronte digitali di DNA attraverso la risoluzione simultanea di lunghezza e variabilità nucleotidica da spettrometria di massa ad alte prestazioni. Hum. Mutat. Google Scholar
- 10. Wiegand, P. e M. Kleiber. 2001. Meno è più – riduzione della lunghezza degli ampliconi STR utilizzando primer riprogettati. Int. J. Legal Med. 114:285-287.Crossref, Medline, CAS, Google Scholar
- 11. Butler, J.M., Y. Shen, e B.R. McCord. 2003. Lo sviluppo di ampliconi STR di dimensioni ridotte come strumenti per l’analisi del DNA degradato. J. Forensic Sci. 48:1054-1064.Crossref, Medline, CAS, Google Scholar
- 12. Dixon, L.A., A.E. Dobbins, H.K. Pulker, J.M. Butler, P.M. Vallone, M.D. Coble, W. Parson, B. Berger, et al. 2006. Analisi del DNA degradato artificialmente usando STRs e SNPs-risultati di un esercizio collaborativo europeo (EDNAP). Forensic Sci. Int. 164:33-44.Crossref, Medline, CAS, Google Scholar
- 13. Hill, C.R., M.C. Kline, J.J. Mulero, R.E. Lagace, C.W. Chang, L.K. Hennessy, and J.M. Butler. 2007. Studio di concordanza tra il kit di amplificazione PCR AmpFlSTR MiniFiler e i kit di tipizzazione STR convenzionali. J. Forensic Sci. 52:870-873.Crossref, Medline, CAS, Google Scholar
- 14. Leibelt, C., B. Budowle, P. Collins, Y. Daoudi, T. Moretti, G. Nunn, D. Reeder, e R. Roby. 2003. Identificazione di una mutazione del sito di legame del primer D8S1179 e la validazione di un primer progettato per recuperare gli alleli nulli. Forensic Sci. Int. 133:220-227.Crossref, Medline, CAS, Google Scholar
- 15. Istituto Nazionale di Giustizia (NIJ). 2000. The future of forensic DNA testing: predictions of the Research and Development Working Group of the National Commission on the Future of DNA Evidence, Washington, D.C. www.ojp.usdoj.gov/nij/pubs-sum/183697.htm.Google Scholar
- 16. Gill, P. 2002. Ruolo del DNA a ripetizione tandem breve nei casi forensi nel Regno Unito – passato, presente e prospettive future. BioTechniques 32:366-372.Link, CAS, Google Scholar
- 17. Gill, P., D.J. Werrett, B. Budowle, e R. Guerrieri. 2004. Una valutazione se gli SNPs sostituiranno gli STRs nei database nazionali del DNA – considerazioni congiunte del gruppo di lavoro sul DNA della Rete Europea degli Istituti di Scienze Forensi (ENFSI) e del Gruppo di lavoro scientifico sui metodi di analisi del DNA (SWGDAM). Sci. Justice 44:51-53.Crossref, Medline, Google Scholar